Поиск сайтов связывания микро РНК в цис-регуляторных последовательностях и в SNP в генах липидного, углеводного обмена, окислительного и противовоспалительного гомеостаза
Aннотация
Актуальность: Полиморфизмы и нарушение профиля экспрессии генов miRNA связывают с системными заболеваниями (аутоиммунные и сердечно-сосудистые заболевания). Цель исследования:Изучение локализации сайтов связывания miRNA с mRNA в цис-регуляторных районах генов и в кодирующих последовательностях ДНК (CDS) ассоциированных в наших исследованиях с ранним и поздним атеросклерозом, а также поиск возможных локализаций сайтов связывания с микроРНК с участками генов (PPARGC1A; LIPC; PON1; APOE; LPL;APOC3; EDN; TNFα;SERPINE1). Материалы и методы:Поиск мотивов гомологичных микро РНК осуществлялся в цис-регуляторных районах изучаемых генов с помощью биоинформационного пакета MEME Suite. Известные miRNA мы взяли из электронной базы данных под названием «miRBase» (адрес сайта http://mirbase.org/). Из базы данных NCBI нами были получены нуклеотидные последовательности цис-регуляторных районов и интронов генов (адрес сайта http://www.ncbi.nlm.nih.gov/) при помощи сочетания скриптов, IFITCH, созданных для автоматизированного получения информации от последовательностей NCBI. Ранее упомянутая нами электронная база данных «miRBase» анализировали автоматически при помощи поиска сайтов связывания в исходной последовательности, при этом мы использовали программу «MScanner». Было зарегистрировано 28645 микроРНК (http://www.mirbase.org). Результаты:Результаты биоинформационного поиска локализации мотивов гомологичных известным микроРНК перед и после гена, а также в кодирующих белок-последовательностях и интронов следующих генов: PON1, APOC3, LIPC, LPL, APOE, PPARGC1A, TNF, EDN, SERPIN показал, что в генах и межгенных пространствах находится большое количество мотивов, гомологичных зрелым микро-РНК. Наибольшее абсолютное количество мотивов локализовано внутри гена PPARGC1A – 22 микроРНК. Однако, если рассматривать относительную частоту локализации микроРНК расчитанную на 100000 п.н., то наибольшее число микроРНК локализовано в гене PON1 и составляет 7.6х10-5. В то время как для гена PPARGС1эта частота составляет 3.2х10-5. Заключение: Таким образом, обнаруженные нами микроРНК локализованные в большом количестве внутри и около изучаемых генов, могут указывать на их функциональную роль в патогенезе атеросклероза.
Ключевые слова: микроРНК, цис-регуляторные последовательности, ранний и поздний атеросклероз, гены: PON1, APOC3, LIPC, LPL, APOE, PPARGC1A, TNF, EDN, SERPIN, сайты связывания
Введение. Полиморфизмы и нарушение профиля экспрессии генов miRNA, в последние годы связывают с различными системными заболеваниями, такими как рак, аутоиммунные и сердечно-сосудистые заболевания. Ранее было показано, что сайты взаимодействия miRNA с mRNA гена расположены на всех участках гена: 3’-нетранслируемом участке (3’UTR), 5'-нетранслируемом участке (5'UTR), белок-кодирующей нуклеотидной последовательности (CDS) [1, 2]. Большинство исследований посвящено изучению взаимодействии miRNA c 3’UTR mRNA. В последние годы показано, что многие miRNA кодируются в межгенных участках ДНК и обнаружены в интронах [3, 4]. Интронные miRNA (in-miRNA) локализованы в интронах белок-кодирующих генов и транскрибируются с ДНК автономно от транскрипции pre-mRNA при наличии собственного промотора, либо вырезаются из pre-mRNA в виде pre-miRNA [5].
За несколько последних десятилетий были получены открытия, коткорые показывают, что РНК может работать не только в качестве функционального посланника, связывающего ДНК и белок, но также может участвовать в регулировании и может организовывать геном, также может регулировать экспрессии генов, а роль РНК непосредственно увеличивается с усложнением (увеличением сложности) организма [6, 7]. Продемонстрирована важность РНК в эпигенетических процессах, контролирующих дифференциацию и развитие. Все эти научные открытия говорят о том, что РНК может играть главную роль в эволюции и онтогенезе человека [8, 9, 10]. Видимо, что и микроРНК играет весомую регуляторную роль в процессе формирования атеросклероза.
Целью данной работы было изучение локализации сайтов связывания miRNA с mRNA в цис-регуляторных районах генов и в кодирующих последовательностях ДНК (CDS) ассоциированных в наших исследованиях с ранним и поздним атеросклерозом, а также поиск возможных локализаций сайтов связывания с микроРНК с участками генов, в котором локализованы изучаемые полиморфизмы.
Материалы и методы. Нуклеотидные последовательности цис-регуляторных районов, CDS последовательностей и интронов генов мы получили из электронной базы данных под названием «NCBI» (адрес сайта http:///www.ncbi.nlm.nih.gov/). Известные микроРНК выгружали из базы данных «miRBase» (http://mirbase.org/), в которой на момент проведения исследования было зарегистрировано 28645 микроРНК человека, далее проводили поиск сайтов связывания с помощью автоматизированной программы «MScanner» [11, 12].
Результаты исследования и их обсуждение. Проведенный нами анализ биоинформации показывает, что в генах и в межгенных пространствах может находиться дстаточно большое количество мотивов, гомологичных зрелым микро-РНК. В таблице 1 представлены результаты биоинформационного поиска локализации мотивов гомологичных известным микроРНК перед и после гена, а также в кодирующих белок-последовательностях и интронов следующих генов: PON1, APOC3, LIPC, LPL, APOE, PPARGC1A, TNF, EDN, SERPIN.
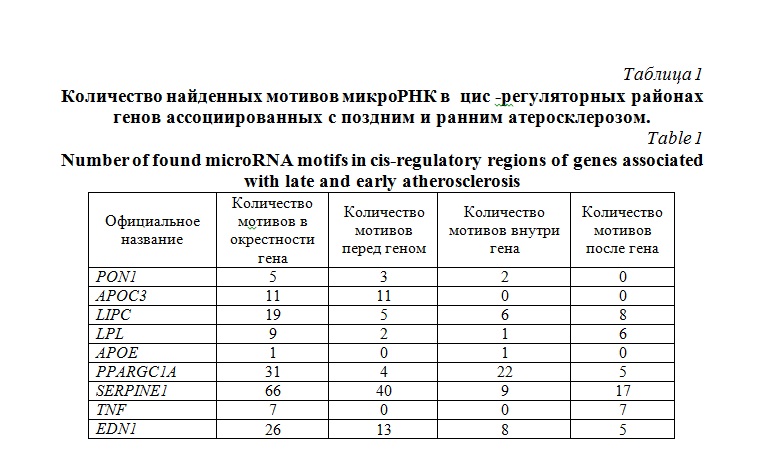
Как видно из представленных результатов наибольшее абсолютное количество мотивов локализовано внутри гена PPARGC1A – 22 микроРНК. Однако, если рассматривать относительную частоту локализации микроРНК расчитанную на 100000 п.н., то наибольшее число микроРНК локализовано в гене PON1 и составляет 7.6х10-5 . В то время как для гена PPARGС1 эта частота составляет 3.2х10-5 . Относительная частота распределения мотивов микоРНК в зависимости от длины гена и изучаемого межгенного участка представлено на рисунке 1.
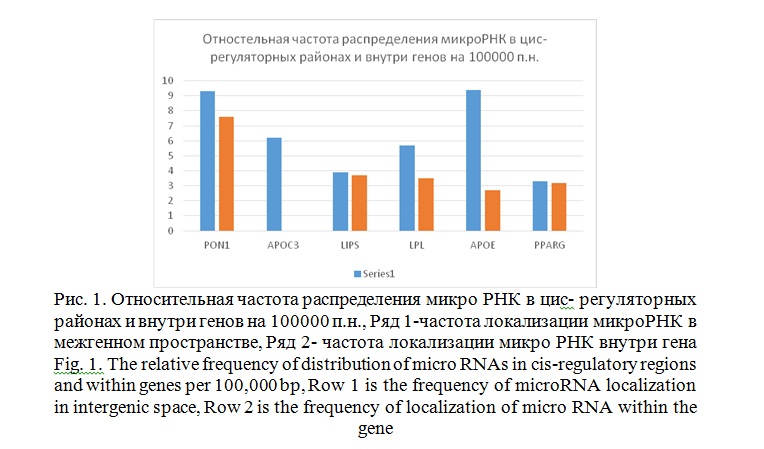
Как видно из представленных результатов, в межгенном пространстве частота локализации микроРНК значительно выше, чем внутри кодирующего участка гена и интронов.
Таким образом, обнаруженные нами микроРНК локализованные в большом количестве внутри и около изучаемых генов, могут указывать на их функциональную роль в патогенезе атеросклероза.
Анализ сайтов связывания различных типов зрелых микроРНК в межгенном пространстве и внутри исследуемых генов представлен в таблице 2
Как видно из таблицы, наиболее часто встречались мотивы, гомологичные, hsa -mir-619 (135 копий), hsa-mir-5096 (91 копия), hsa-mir-1273 (30 копий) hsa-mir 5585 (26 копий), has-mir-548 (13 копий), mir-1285 (12 копии), hsa-mir-5095 (10 копии), hsa-mir-1303 (6 копий). Большинство последовательностей встречалось в двух копиях: hsa-mir-3674 (2 копии), hsa-mir-297 (2 копии), hsa-mir-3622 (2 копии), hsa-mir-1972 (2копии), hsa-mir-4687 (2 копии), hsa-mir-4277 (2 копии), hsa-mir-3116 (2копии), hsa-mir-6867 (2копии), hsa-mir-8062 (2копии).
Ранее было показано, что сайты связывания miR-5096 локализуются на 832 таргетных матричных РНК [13]. Возможно, такое достаточно большое количество сайтов связывания говорит нам о функциональной роли miR-5096 в качестве координатора экспрессии больших генных ансамблей. Ранее было обнаружено высокое копировальное качество генов hsa -mir-619 и hsa-mir-5096 , обнаружили это при проведении анализа распространения микро РНК по кругу от оси генов соматоропина (GH1, GHRH, SST, и IGF1) [14].
При гипертонии была обнаружена циркулирующая микроРНК hsa-circ-0005870 , которая при построении авторами генных сетей по KEGG-пути включила микроРНК hsa-miR-6807-3p, hsa-miR-5095, hsa-miR-1273g-3p, hsa-miR-5096, hsa-miR-619 -5р и их соответствующие мРНК [15, 16].
Определение miR-126, miR-155, miR-221, miR-222 в группах больных с гипертонической болезнью и ишемической болезнью сердца подтверждает корреляцию изучаемых микроРНК с исследуемыми заболеваниями сердечно-сосудистой системы [17].
Особый интерес представляют микроРНК сайты связывания, которых, были представлены в единичных случаях это: miR-8096, miR-3116-2, miR-4277, miR-566, miR-5009, miR-8069-2, miR-3133, miR-7113, miR-1304, miR-5708, miR-4496, miR-649, miR-6847, miR-1304, miR-3834, miR-4687, miR-3674. Возможно, эти микроРНК принимают важное участие в экспрессии генов, рядом с которыми они локализованы на разных этапах онтогенеза, в разных тканях. Очевидно, не все они будут связаны с развитием атеросклероза, но они представляют большой потенциал для изучения.
Цис-регуляторные районы генов, вовлеченных в процессы развития атеросклероза содержат таргетные мишени генов микро РНК mir-548 hsa -mir-619, hsa-mir-1273, hsa-mir-5096, has- mir-548, hsa-mir 5585, has-mir-5069.
Полиморфный локус гена PON1(Gln192Arg) расположен рядом с микро-РНК hsa-mir-6867 и hsa-mir-4277. Полиморфный локус гена PPARGC1A Gly482Ser расположен рядом с микро-РНК hsa-mir-297. Внутри гена PPARGC1A нами была обнаружена локализация сайта связывания с miR-297. С другой стороны, показано, что микроРНК управляет непосредственно эндотелиальной клеткой, а также клетками гладких мышц кровеносных сосудов и функциями макрофага, и таким образом регулирует прогрессию атеросклероза, в частности miR-297 изменяет экспрессию гена эндотелиального фактора роста сосудов VEGFA [18, 19], который, в свою очередь, ассоциирован с атеросклерозом (OMIM: 192240) [20].
Заключение. Таким образом, описанные нами новые микроРНК могут реально быть вовлечены в патогенез атеросклероза. Перспективы дальнейших исследований в этой области связаны с разработкой диагностических методов, развитием новых подходов лечения этих заболеваний на основе miRNA, предсказыванием и прогнозированием эффективности терапии.
В отношении данной статьи не было зарегистрировано конфликта интересов.




















Список литературы