Роль метилирования ДНК при раке легкого (обзор)
Aннотация
Актуальность: Рак легкого на протяжении десятилетий остается трудно поддающимся лечению заболеванием, несмотря на усилия онкологов и фармацевтических компаний. Одной из причин этого является неоднородность генетических профилей опухолей. В последние годы интерес исследователей привлекают эпигенетические изменения внутри опухоли, в частности, метилирование ДНК. В данной работе рассматриваются отличительные особенности основных подтипов рака легкого, обсуждается роль метилирования ДНК при немелкоклеточном и мелкоклеточном раке легкого, а также его роль в ответе опухоли на терапию. Цель исследования:Обобщение результатов анализа метилирования ДНК при раке легкого для лучшего понимания биологической природы онкогенеза данного заболевания, использования выявленных биомаркеров для ранней диагностики патологии и оценки прогноза пациента. Материалы и методы:Анализ литературы проводился в базах данных PubMed, Google Scholar за период с 1995 по 2022 гг. Результаты:Показано, что паттерн метилирования ДНК меняется под воздействием курения, причем изменения сохраняются даже после его прекращения.Выявлены аберрантно метилированные гены, которые могут служить биомаркерами для скрининга рака легкого на ранних или поздних стадиях. К числу таких генов относятся: H19, HOXA3/HOXA4, RUNX3, BRICD5, PLXNB2, RP13, DIABLO, RASSF1A, SHOX2, EZH2, NEUROD1 и другие.Более того, обнаружено, что статус метилирования отдельных областей ДНК при мелкоклеточном раке легкого коррелирует с ответом опухоли на химиотерапию и лучевую терапию. Заключение:Изменения в профилях метилирования ДНК индивидов могут служить маркерами для ранней диагностики заболевания, использоваться в качестве мишеней для терапевтического воздействия, а также для оценки прогноза пациента
Введение. Рак легкого (РЛ) является одним из самых распространенных онкозаболеваний и ведущей причиной смертности от них во всем мире: по оценкам Международного агентства по изучению рака, в 2020 г. было зарегистрировано 2,2 миллиона новых случаев заболевания и 1,8 миллиона случаев смерти [1]. Основываясь на гистологии опухоли, выделяют два основных подтипа РЛ – немелкоклеточный (НМРЛ) и мелкоклеточный (МРЛ).
НМРЛ составляет приблизительно 85% всех случаев РЛ и представлен несколькими типами, в том числе аденокарциномой легкого (наиболее распространенный тип РЛ), плоскоклеточным раком и крупноклеточной карциномой.
В ходе геномных исследований, проведенных в рамках проекта Clinical Lung Cancer Genome Project, было выявлено, что для различных типов НМРЛ характерны специфичные изменения драйверных генов. В частности, при аденокарциноме легкого часто обнаруживаются активирующие мутации в гене рецептора эпидермального фактора роста EGFR и перестройки EML4-ALK. Кроме того, при данном типе РЛ идентифицированы мутации в генах LKB1/STK11, CDKN2A, NF1, KEAP1,KRAS, амплификация MET и перестройки ROS1 и RET [2]. При плоскоклеточном РЛ, напротив, мутации EGFR или перестройки EML4-ALK наблюдаются редко. Для этого типа РЛ характерны изменения в генах рецепторных тирозинкиназ, DDR2 и генах рецепторов фактора роста фибробластов, а также инактивирующие мутации в CDKN2A, PTEN, KEAP1, NFE2L2 и RB1 [3]. Перечень генов, мутации в которых свойственны для аденокарциномы легкого и плоскоклеточного РЛ, представлен на рисунке.

На долю МРЛ в глобальном масштабе приходится около 15% всех случаев РЛ, причем ежегодно диагностируется около 250000 новых случаев [6]. МРЛ представляет собой агрессивную нейроэндокринную опухоль высокой степени злокачественности, которая отличается исключительно высокой скоростью пролиферации и склонностью к раннему метастазированию. На момент постановки диагноза лишь у трети пациентов заболевание будет выявляться на ограниченной стадии. Для остальных пятилетняя выживаемость составляет менее 1%. Стоит отметить, что наиболее часто метастазы наблюдаются в контралатеральном легком, мозге, печени, надпочечниках и костях [7].
Для МРЛ показана выраженная связь с курением – только в 2% случаев МРЛ возникает у никогда не куривших [6]. Значительную роль в развитии МРЛ канцерогенов табачного дыма подтверждает мутационный профиль данного типа опухолей, характеризуемый множеством нуклеотидных перестроек. [7, 8]. Наиболее распространенным генетическим изменением при МРЛ является биаллельная инактивация генов-супрессоров опухолевого роста TP53 и RB1. Также часто выявляется увеличение числа копий генов, кодирующих члены семейства MYC (протоонкогены MYC, MYCL и MYCN), генов ферментов, участвующих в ремоделировании хроматина, рецепторных тирозинкиназ и их нижестоящих эффекторов, повышенный уровень экспрессии ингибиторных белков семейства Notch [6]. В таблице 1 отображены результаты масштабного исследования генома МРЛ.
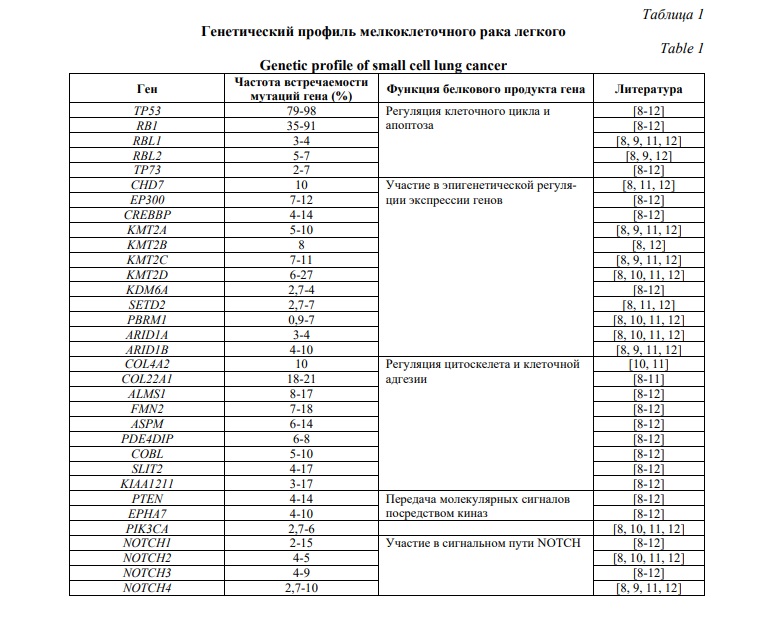
Помимо этого, исследования последних лет позволили выделить четыре подтипа МРЛ, основываясь на различиях в экспрессии регуляторов транскрипции ASCL1 (achaete-scute homologue 1), NEUROD1 (neurogenic differentiation factor 1), YAP1 (yes-associated protein 1) и POU2F3 (POU class 2 homeobox 3) [13]. Впоследствии подтип МРЛ, определяемый экспрессией YAP1, не подтвердился [14]. Однако Gay с соавторами предложили ввести четвертый подтип МРЛ и назвать его воспалительным, поскольку для данной группы опухолей была характерна экспрессия генов многочисленных иммунных контрольных точек и человеческих лейкоцитарных антигенов (HLA) [15].
Значение генетических изменений в злокачественной трансформации опухоли не вызывает сомнений. Тем не менее, в настоящее время признается существенной роль в канцерогенезе и эпигенетических механизмов контроля работы генома, которые включают в том числе специфические изменения метилирования ДНК, посттрансляционную модификацию гистоновых белков и изменения профиля экспрессии микроРНК.
Цель исследования. Обобщение результатов анализа метилирования ДНК при РЛ для лучшего понимания биологической природы онкогенеза данного заболевания, использования выявленных биомаркеров в ранней диагностике патологии и оценке прогноза пациента.
Метилирование ДНК у больных РЛ
Одной из наиболее изученных эпигенетических модификаций геномной ДНК является метилирование. Считается, что изменение профиля метилирования ДНК является одним из самых ранних молекулярных маркеров онкологических заболеваний человека [16, 17, 18]. Геном опухолевых клеток обнаруживает глобальное гипометилирование и региональное гиперметилирование, особенно в промоторных CpG-островках генов-супрессоров опухолевого роста [16]. Метилирование промоторных CpG-островков влияет на связывание транскрипционных факторов и таким образом может подавлять экспрессию совместно действующих генов, большинство из которых являются генами-супрессорами опухолевого роста, что отражает значение данного процесса в онкогенезе [19].
Очень часто исследования, посвященные метилированию ДНК у больных РЛ, проводятся на образцах периферической крови, ввиду значительно большей доступности материала. Несмотря на то, что паттерн метилирования ДНК в крови может и не отражать метилирование в ткани, он может быть показателем серьезных иммунологических изменений или изменений эпигенетической программы, значимых в процессе развития заболевания.
Значительное количество работ по анализу метилирования при РЛ показало прямую связь этого явления с курением [20, 21, 22]. Так, в метаанализе, включавшем около 16000 образцов ДНК курящих и некурящих индивидов, было выявлено 2623 дифференциально метилированных CpG сайта, связанных с курением табака. Для генов, в которых локализованы данные CpG, ранее в полногеномных исследованиях были показаны ассоциации с развитием вызванных курением патологий легких, сердца, воспалительных и онкологических заболеваний [23]. Кроме того, сравнение профилей экспрессии у никогда не куривших и бросивших курить обнаружило 185 дифференциально метилированных CpG сайтов, что отражает устойчивое изменение паттерна метилирования у индивидов, сохраняющееся даже после прекращения воздействия курения.
Связь метилирования ДНК и статуса курения была показана и в других работах. Baglietto с соавт. в результате полногеномного эпигенетического исследования (EWAS) обнаружили 6 CpG сайтов, гипометилирование которых ассоциировано с развитием РЛ, в том числе описанные ранее сайты в генах AHRR (репрессор арилгидрокарбонового рецептора) и F2RL3 (белок 3, подобный рецептору фактора коагуляции II (тромбина)) [20, 22]. Интересно, что уровень метилирования в изученных образцах изменялся градиентно от курящих на момент забора материала к прекратившим курить и никогда не курившим, и был тем выше, чем раньше пациент прекращал курить [22].
В более поздней работе Sandanger с соавторами выявили 25 CpG сайтов, ассоциированных с развитием РЛ, причем после введения поправки на воздействие курения статистически значимая ассоциация сохранилась лишь для двух сайтов, таким образом, они рассматриваются как несвязанные с курением, тогда как остальные 23 CpG сайта связаны с развитием РЛ у курящих. В целом, показано, что ассоциация для 23 CpG сайтов была выше в гистологических типах РЛ, наиболее коррелирующих с курением. Сравнение экспрессионных профилей показало различные пути обогащения таких сайтов, т.е. CpG сайты, связанные и не связанные с курением, по-видимому, вовлечены в различные биологические процессы. Нужно отметить, что в данном исследовании выявленные ранее CpG сайты в генах AHRR и F2RL3 оказались в числе тех, что были ассоциированы с курением [24].
В исследовании Battram с соавторами также было выявлено 16 CpG сайтов, ассоциированных с риском развития РЛ, однако после менделевской рандомизации данная ассоциация существенно снизилась [25]. В частности, полученные ими результаты не согласуются с предыдущими данными, свидетельствующими о том, что CpG сайты в генах AHRR и F2RL3 опосредуют более 30% эффекта курения на развитие РЛ [20]. Можно предположить, что наблюдаемая ранее ассоциация изменений метилирования ДНК с риском развития РЛ возникает как результат внесения поправок на сведения о курении, полученные от пациентов [26, 27]. К таким сведениям относятся количество выкуренных сигарет в течение жизни (более или менее 100 сигарет), статус курения на момент исследования, регулярность курения. При применении в EWAS-исследованиях более строгих поправок, данные ассоциации также пропадают.
Курение значительно изменяет паттерн метилирования ДНК, который сохраняется даже после его прекращения, и, по-видимому, это является одним из механизмов, объясняющих негативное воздействие его на здоровье человека. Тем не менее, наблюдаемая ассоциация между метилированием ДНК и развитием РЛ может отражать независимые влияния курения на метилирование и на развитие РЛ.
Выявление изменений метилирования ДНК, не связанных с активным курением, позволит обнаружить новые биологические пути, функционирование которых значимо в контексте развития РЛ. Тем не менее, на сегодняшний день таких исследований очень мало.
В одном из них выявлены 151 CpG сайт, связанный с курением, и 3806 CpG сайтов, не связанных с курением, и далее проведен анализ ассоциации их со смертностью от РЛ. CpG сайты, связанные с курением, оказались ассоциированы со смертностью от РЛ с большей значимостью [28]. Результаты подобных исследований могут способствовать более точному отбору популяции для скрининга РЛ.
При изучении роли в развитии РЛ сайленсинга генов посредством их метилирования выявляется множество ассоциированных CpG сайтов в различных локусах, и анализ такого объёма данных требует значительных ресурсов, в связи с чем целесообразнее было бы определять основные драйверные гены, изменение профиля метилирования которых вовлечено в онкогенез. Для выявления таких ключевых генов Zhang с соавторами при исследовании образцов опухолевой и прилежащей к ней нормальной ткани легкого проводили сопоставление данных по метилированию с экспрессионными данными. В результате данного анализа ими было установлено 30 драйверных генов с измененным профилем метилирования, ассоциированных с развитием РЛ у некурящих индивидов [29].
В другом недавнем исследовании, проведенном на когорте CLUE II (образцы крови пресимптоматических индивидов, забранные в 1989 г. в США), авторы проводили поиск отдельных CpG сайтов и дифференциально метилированных регионов (ДМР), ассоциированных с развитием РЛ независимо от курения [30]. Выборка включала две группы индивидов: тех, кто позднее заболел РЛ, и тех, у кого данное заболевание выявлено не было. Оценку роли участков метилирования ДНК в развитии РЛ проводили, опираясь на данные об известных изменениях паттерна метилирования, связанных с курением, с использованием индекса пачка/лет.
Авторами выявлено 16 CpG сайтов и 40 ДМР, ассоциированных с развитием РЛ, причем значение OR было выше для бывших курильщиков и для пациентов с МРЛ, по сравнению с пациентами с НМРЛ. Данные участки с измененным метилированием локализованы в генах H19, HOXA3/HOXA4, RUNX3, BRICD5, PLXNB2, RP13 и DIABLO, для которых ранее показана ассоциация с РЛ [31, 32, 33].
Продукты генов гомеобоксных белков HOXA являются факторами транскрипции, регулирующими экспрессию генов, морфогенез и дифференцировку тканей. Известно, что экспрессия фактора транскрипции HOXA3 снижена при аденокарциноме и плоскоклеточном РЛ. Кроме того, показано, что у курящих индивидов уровень HOXA3 заметно ниже, чем у некурящих [34]. Сравнение паттерна метилирования в опухоли и прилегающей нормальной ткани при аденокарциноме выявило ассоциацию с ДМР в области гена HOXA3 [35]. Ген другого фактора транскрипции RUNX3 также является опухолевым супрессором. Гиперметилирование промотора данного гена ассоциировано со снижением выживаемости при РЛ [36]. Кодируемый геном DIABLO митохондриальный белок является проапоптотическим белком и обеспечивает активацию каспаз путем связывания с белками-ингибиторами апоптоза. Различные исследования свидетельствуют об участии данного белка в онкогенезе. Одними авторами предполагается, что повышенная экспрессия его повышает чувствительность опухолевых клеток к апоптозу, предотвращая таким образом опухолевую прогрессию [37]. Другие исследователи рассматривают гиперэкспрессию DIABLO необходимым звеном в запуске опухолевой метаплазии [38].
Также известна роль длинной некодирующей РНК H19 в развитии РЛ. Угнетение экспрессии Н19 приводит к подавлению роста, миграции и инвазии при НМРЛ. Подавление её импринтинга коррелирует с тотальным деметилированием и ассоциировано с трансформацией нормальной ткани в НМРЛ [39, 40].
В вышеописанной работе Zhao с соавторами была выявлена ассоциация гиперметилирования многих CpG сайтов в области гена H19 с развитием РЛ. Ранее же в других исследованиях была показана гиперэкспрессия Н19 в опухоли РЛ, которая коррелирует с гипометилированием промоторных CpG [41].
Интересно, что метилирование региона H19 подвержено воздействию разнообразных модифицирующих влияний, начиная с внутриутробного периода и раннего детства, что может говорить о возможной роли в данном процессе внешних факторов. Это может иметь значение при изучении различных механизмов онкогенеза, его этапов и ключевых элементов.
Результаты многих исследований РЛ в последнее время демонстрируют снижение экспрессии вследствие гиперметилирования ряда генов супрессоров опухолей, в т.ч. CDKN2A, APC, RARβ, MGMT, DAPK, F2RL3 и RASSF1A [42, 43, 44]. Ген RASSF1A, кодирующий белок семейства 1А, содержащий Ras-ассоциированный домен, является одним из наиболее тщательно изученных супрессоров опухолевого роста, функционально он участвует в пролиферации клеток, миграции и онкогенезе [45]. Онкоген SHOX2 (гомеобокс содержащий ген низкорослости 2) является регулятором клеточной пролиферации и апоптоза и индуктором эпителиально-мезенхимального перехода [46]. Помимо онкогенных функций SHOX2 также играет значительную роль в процессах эмбрионального развития и дифференцировки клеток сердечно-сосудистой системы и в развитии скелета [47, 48]. Важно отметить, что часто в ткани РЛ выявляется метилирование как гена RASSF1A, так и SHOX2 [49, 50, 51]. Ряд исследований показали, что сочетанное метилирование генов RASSF1A и SHOX2, выявленное в различных типах образцов, включая бронхоальвеолярный лаваж, сыворотку, жидкость из плевральной полости, образцы асцитической жидкости, клетки и лимфатические узлы, продемонстрировало чрезвычайно высокую чувствительность и специфичность при РЛ [49-53]. Имеющиеся данные убедительно свидетельствуют о том, что метилирование RASSF1A и SHOX2 является критическим событием в онкогенезе и прогрессировании РЛ, и эти модифицированные гены могут служить потенциальной парой биомаркеров для скрининга РЛ на ранних или поздних стадиях [42, 48, 51, 54, 55].
Метилирование ДНК при МРЛ
Глобальных исследований метилирования ДНК, посвященных исключительно МРЛ, существенно меньше, чем для НМРЛ, и они проведены на ограниченном числе образцов [56, 57]. В одном из первых анализов профиля метилирования при МРЛ [56] было идентифицировано 73 гена, аберрантно метилированных в более чем 77% первичных опухолей. Анализ генных онтологий показал значительное обогащение метилированных генов, функционирующих как транскрипционные факторы и участвующих в процессах нейрональной дифференциации. Анализ опухоль-специфических метилированных областей выявил обогащение сайтов связывания нескольких транскрипционных факторов нейрональных клеток, включая NEUROD1, HAND1, ZNF423 и REST. Авторы предполагают два вероятных механизма нарушения дифференцировки нейроэндокринных клеток, что стимулирует клетки-предшественники опухоли к трансформации в МРЛ. Первый – инактивация путем метилирования транскрипционных факторов, необходимых для правильной дифференцировки клеток, второй – функциональная инактивация путем метилирования соответствующих ДНК-связывающих участков [56].
Авторы другого исследования, в ходе которого был проведен полногеномный анализ метилирования ДНК в легочной ткани больных МРЛ, обнаружили повышенную экспрессию при МРЛ гистоновой метилтрансферазы EZH2, которая коррелирует с метилированием промоторов множества генов при различных типах опухолей и ассоциирована с плохим прогнозом. В экспериментах на клеточных линиях показано, что ингибирование EZH2 приводит к подавлению роста клеток опухоли, что делает данный белок потенциальной терапевтической мишенью при МРЛ [57]. Кроме того, выявлен сайленсинг других генов, в т.ч. транскрипционных факторов NEUROD1, TCF2 (HNF1B), REST, бета-рецептора ретиноевой кислоты RARB, BCL2 и RASSF1A.
Также результаты данной работы продемонстрировали гипометилирование большинства CpG в первичных опухолях МРЛ по сравнению с образцами здорового легкого. Было показано, что гиперметилированные CpG сайты локализованы преимущественно в области 500 пн выше сайта старта транскрипции, тогда как гипометилированные распределены по всей промоторной области. Причем, обнаружилось неоднородное распределение и самих наиболее гипометилированных CpG сайтов в зависимости от их влияния на экспрессию гена: CpG, ассоциированные с повышенной экспрессией гена, локализованы ниже, а CpG, ассоциированные с сайленсингом гена, – выше старта транскрипции [57].
У многих форм злокачественных новообразований у человека описан так называемый CIMP-фенотип (CpG-island methylator phenotype), который характеризуется одновременным метилированием многих CpG островков промоторной области генов, в нормальной ткани никогда не метилированных. В исследовании Saito с соавторами проведена стратификация образцов МРЛ в соответствии со статусом CIMP. Оказалось, что пациенты с CIMP-положительными опухолями имели худший прогноз, чем пациенты с CIMP-отрицательным заболеванием [58]. Эти данные предполагают, что клинически значимые подтипы МРЛ определяются паттернами метилирования ДНК, что согласуется с наблюдениями среди других эпитипов карциномы легких [6, 59].
Роль метилирования ДНК при МРЛ в ответе опухоли на химиотерапию и лучевую терапию
Одна из особенностей МРЛ состоит в том, что у большинства пациентов наблюдается хороший первоначальный ответ на химиотерапию и лучевую терапию, однако в течение 6-12 месяцев почти у всех больных развивается рецидив с переходом заболевания в резистентную форму [60]. Несмотря на все большее количество доказательств роли эпигенетических факторов в развитии МРЛ, понимание их влияния на ответ опухоли на лечение остается ограниченным. Чтобы получить представление о том, как метилирование ДНК может влиять на ответ опухоли на лечение при МРЛ, Krushkal с соавторами провели полногеномный анализ метилирования ДНК на 66 линиях клеток МРЛ человека [61]. Ими были изучены корреляции метилирования ДНК при МРЛ с чувствительностью к противоопухолевым препаратам.
Потенциально важная ассоциация наблюдалась для гена экзонуклеазы TREX1. Повышенное метилирование и сниженная экспрессия TREX1 были ассоциированы с чувствительностью к некоторым ингибиторам киназы Aurora и ряду других препаратов. По сравнению с клеточными линиями других типов рака, TREX1 характеризовался сниженной экспрессией мРНК и повышенным метилированием вышележащей области при МРЛ, что предполагает наличие возможной связи с чувствительностью МРЛ к ингибиторам киназы Aurora. Krushkal с коллегами также идентифицировали несколько дополнительных корреляций, указывающих на потенциальные механизмы химиочувствительности. Ассоциация ответа на ингибиторы киназы Aurora и другие химиотерапевтические агенты обнаружена также с метилированием генов CEP350, MLPH, EPAS1, KDM1A, EZH2 и EPHA2 [61] (Табл. 2).

В недавнем исследовании Zhai с соавторами продемонстрировали роль гена каталитической субъединицы теломеразы человека (hTERT) в развитии МРЛ. Ими было показано, что метилирование промотора данного гена способствовало прогрессированию МРЛ и резистентности клеток опухоли к лучевой терапии [62]. Сверхэкспрессия hTERT приводила к увеличению транскрипции ряда маркеров эпителиально-мезенхимального перехода, в том числе OCLN, JUP и ZEB, и, таким образом, способствовала миграции клеток МРЛ. Также было выявлено, что повышение экспрессии hTERT влекло повышение экспрессии EZH2, за счет чего усиливалась пролиферация клеток МРЛ.
Другой эпигенетический терапевтический подход к лечению МРЛ открывают исследования по ингибированию лизин-специфической деметилазы 1 (LSD1) [63, 64]. LSD1 представляет собой белок, содержащий флавин и функционирующий как гистондеметилаза и корепрессор транскрипции [65]. Данный белок является широко изученным членом семейства LSD, и повышенная экспрессия его показана при многих видах рака, включая подтипы МРЛ. Белок LSD1 деметилирует гистон H3 по четвертому лизину, что приводит к подавлению транскрипции, или деметилирует данный гистон по девятому лизину, что имеет противоположный эффект [66].
Augert с соавторами показали, что ингибирование LSD1, опосредованное его селективным ингибитором ORY-1001, приводит к активации сигнального пути NOTCH и подавляет экспрессию транскрипционного фактора ASCL1, что тормозит прогрессирование МРЛ [64]. Активация NOTCH, наряду с ингибированием ASCL1, имеет терапевтический потенциал при МРЛ, поскольку высокий уровень экспрессии NOTCH и низкий уровень ASCL1 также подавляет дифференцировку нейроэндокринных клеток. Ингибитор ORY-1001 продемонстрировал эффективность на хеморезистентных моделях ксенотрансплантатов, полученных от пациентов с МРЛ.
Mohammad с коллегами сообщили о другом селективном, биодоступном при пероральном введении сильнодействующем ингибиторе LSD1 (GSK2879552), который проявляет противоопухолевые свойства на клеточных линиях МРЛ и моделях опухолей [63]. Эффективность GSK2879552 зависела от статуса метилирования ДНК. Авторами был подобран набор из 45 дифференциально метилированных зондов, способных предсказывать чувствительность МРЛ к данному препарату. Статус гипометилирования этого набора зондов позволяет стратифицировать пациентов с МРЛ, которые могут реагировать на ингибирование LSD1 с помощью GSK2879552 [63, 67].
Заключение. В целом, исследования метилирования при МРЛ призваны прояснить биологическую природу онкогенеза при данном заболевании. Различия в профилях метилирования ДНК, выявляемые у разных индивидов, могут отражать различную генетическую предрасположенность к РЛ. Это может быть обусловлено некоторыми индивидуальными различиями в системе детоксикации ксенобиотиков, в том числе канцерогенов, либо различиями в функционировании систем репарации, которые должны восстанавливать возникающие вследствие стрессовых влияний нарушения. С другой стороны, различия в метилировании могут отражать и эффект от воздействия различных факторов, поступающих из внешней среды.
С учетом того, что для эпигенетических маркеров характерна достаточная пластичность, любые изменения в метилировании ДНК, ассоциированные с развитием РЛ, могли бы быть использованы в качестве мишеней для терапевтического воздействия. Полученные в результате множества исследований результаты дают надежды на расширение арсенала химиотерапевтических препаратов. Помимо этого, немаловажным аспектом является потенциальное применение выявляемых маркеров для ранней диагностики заболевания и оценки прогноза пациента.
Тем не менее, возникают определенные трудности с правильным определением и точным анализом эпигенетических маркеров. Данные маркеры чувствительны к различным, в том числе обратным воздействиям в процессе онкогенеза. Кроме того, иногда затруднительно их отделить от влияния социо-экономических факторов. Таким образом, для дальнейшего анализа требуется детальное изучение биологических путей и определение всех ключевых их компонентов.
Информация о финансировании
Работа проведена при финансовой поддержке Санкт-Петербургского государственного университета (проект № 93025749) и мегагранта Правительства Российской Федерации № 075-15-2021-595.




















Список литературы