Частота, спектр и функциональное значение мутаций в гене MIR-142 при диффузной В-клеточной крупноклеточной лимфоме
Aннотация
Актуальность:MIR-142 является единственным геном микроРНК человека, в котором при диффузной В-клеточной крупноклеточной лимфоме (ДВККЛ) с высокой частотой выявляются мутации. Ранние исследования нарушений в данном гене были основаны на анализе нескольких десятков биообразцов лимфомы и не уделяли внимания оценке функционального эффекта выявленных изменений. Цель исследования:Описать частоту и спектр мутаций в гене MIR-142 на крупной выборке образцов опухолевой ткани пациентов с ДВККЛ, а также провести анализ in silico их функционального значения. Материалы и методы:Проведено прямое секвенирование по Сенгеру гена MIR-142 в 123 образцах ДНК, выделенной из срезов FFPE-блоков биоптатов опухолевых лимфоузлов пациентов с ДВККЛ. Предсказание in silico генов-мишеней для каждой из «ключевых» последовательностей микроРНК выполнено с помощью онлайн-инструмента miRDB. Для определения функции генов-мишеней микроРНК проведен анализ обогащения терминов генных онтологий молекулярных функций и биологических процессов с помощью PANTHER на базе биоинформационного ресурса Gene Ontology. Для предсказания вторичной структуры «шпильки» микроРНК и расчета ее минимальной свободной энергии (dG) использовался UNAfold web server. Результаты:Частота мутаций в группе исследования составила 16,7%, в трех случаях имели место множественные однонуклеотидные замены в последовательности MIR-142. Всего выявлено 24 типа однонуклеотидных замен, транзиции преобладали над трансверсиями. Анализ распределения мутаций по последовательности гена MIR-142 показал, что лишь 15% было локализовано в «ключевых» областях, большая же часть – за ее пределами: в последовательности зрелых цепей (30%), предшественника (40%) или первичного транскрипта (10%) микроРНК. Анализ in silico влияния замен, расположенных в «ключевой» последовательности, на взаимодействия микроРНК-мРНК показал, что все три выявленные в группе исследования мутации приводят к существенному изменению набора регулируемых генов. Наиболее выраженные изменения спектра регулируемых генов установлены для n.68G/A. Практически все описанные нами мутации в той или иной степени влияли на структуру и термостабильность микроРНК «шпильки». Заключение:Полученные данные свидетельствуют о существенном влияние мутаций в «ключевой» последовательности на переключение генов-мишеней зрелых цепей изучаемой микроРНК. Необходимы дальнейшие функциональные исследования для подтверждения влияния предсказанных изменений термодинамической стабильности и вторичной структуры на биогенез зрелых цепей микроРНК
Ключевые слова: диффузная В-клеточная крупноклеточная лимфома, регуляция микроРНК, экспрессия генов, miR-142, мутационный профиль, высокопроизводительное секвенирование
Введение. Диффузная крупноклеточная В-клеточная лимфома (ДВККЛ) относится к группе орфанных лимфопролиферативных новообразования и представляет собой агрессивную опухоль. На ее долю приходится до 60% В-клеточных и 40% всех случаев неходжкинских злокачественных лимфом. Последние десятилетия в Российской Федерации и в мире в целом наблюдается неуклонный рост заболеваемости ДВККЛ. Несмотря на внедрение в протоколы ведения пациентов с данным вариантом опухоли инновационных таргетных терапевтических опций, первичная резистентность к лечению или рецидивы заболевания констатируются у 30-40% пациентов, что связано с крайне неблагоприятным прогнозом для жизни. Все это обусловливает необходимость исследований, направленных на более глубокое понимание механизмов лимфомагенеза и молекулярной гетерогенности ДВККЛ [1].
Анализ данных полученных в рамках крупных проектов по изучению опухолевых геномов, позволил идентифицировать тысячи мутаций в генах, кодирующих белки и ассоциированных со злокачественной трансформацией [2]. В том числе, в последние годы исследователями приложены большие усилия по характеристике генома и транскриптома ДВККЛ, включая анализ на уровне отдельных клеток [3]. Полученные данные позволили описать молекулярно-генетически различные подтипы заболевания и разработать новые прогностические модели, основанные на мутационном ландшафте [4-10], картине экспрессии генов в опухоли [11, 12, 13] или характеристике ее микроокружения [14, 15].
Вместе с тем современные знания об изменениях в нуклеотидной последовательности генома вне локусов расположения белок-кодирующих генов, составляющих около 2% от всего генома человека, в частности, в областях, которые несут информацию о некодирующих РНК, все еще ограничены. При этом некодирующие РНК, в частности, микроРНК регулируют экспрессию белок-кодирующих генов на посттранскрипционном уровне, выступают в качестве онкомиров или онкосупрессоров и считаются важными участниками онкогенеза при ДВККЛ [16, 17].
Анализ Urbanek-Trzeciak M.O. et al. (2020) крупных наборов данных секвенирования, полученных в рамках проекта The Cancer Genome Atlas (TCGA), позволил выявить и охарактеризовали более 10 000 мутаций в генах микроРНК при 33 типах новообразований [18], каждая из которых потенциально может влиять на процессинг или функционирование кодируемых ими молекул [19].
Биогенез микроРНК является сложным процессом и включает в себя два основных этапа. На первом из них длинный первичный транскрипт микроРНК (pri-miRNA), транскрибируемый из гена микроРНК, расщепляется ферментным комплексом Drosha/DGCR8 с образованием молекулы-предшественницы – пре-микроРНК (pre-miRNA). Далее пре-микроРНК подвергается вторичному расщеплению ферментом Dicer, что приводит к высвобождению дуплекса микроРНК. В последующем одна зрелая нить этого дуплекса, называемая направляющей, включается в эффекторный РНК-индуцированный сайленсинг-комплекс (RISC) вместе с белком Argonaute. RISC оказывает свое влияние на мРНК генов посредством комплементарного связывания последовательностей микроРНК и мРНК, что приводит к подавлению трансляции или деградации мРНК, и, следовательно, нарушению синтеза белка [17, 20].
Одним из важных результатов работы с данными проекта TCGA стал тот факт, что MIR-142, кодирующий зрелые функционально активные цепи miR-142-3р и miR-142-5р, является единственным геном микроРНК человека, который рекуррентно мутирует при гемобластозах: остром миелоидном лейкозе, хроническом лимфолейкозе и различных типах В-клеточных лимфом [18]. Вместе с тем, все проведенные в настоящее время исследования мутаций в данном гене основаны на анализе нескольких десятков биообразцов опухолей. При этом в работах практически не уделено внимания оценке функционального эффекта выявленных изменений.
Цель исследования. Цель настоящего исследования заключалась в описании частоты и спектра мутаций в гене MIR-142 на крупной выборке образцов опухолевой ткани пациентов с ДВККЛ, а также анализе in silico их функционального значения.
Материал и методы исследования. Были проанализированы 125 образцов ДНК, выделенной из срезов FFPE-блоков (фиксированных формалином, залитых парафином) биоптатов опухолевых лимфоузлов пациентов с ДВККЛ, диагностированных в Городском гематологическом центре г. Новосибирска в период с 2007 по 2022 гг. Исследование было одобрено Комитетом по биоэтике Новосибирского государственного медицинского университета и проводилось в соответствии с Хельсинкской декларацией. От всех пациентов, включенных в это исследование, было получено добровольное информированное согласие.
Ген MIR-142 амплифицировали описанным ранее методом [21] с использованием следующих праймеров: 5'-CTCACCTGTCACACGAGGTC-3’ и 5'-CTCTTGAGCAGGAGGTCAGG-3’. Получали продукт длиной 231 п.н., включающий всю последовательность первичного транскрипта miR-142 вместе с фланкирующими областями длиной около 25 н.п. Продукты ПЦР очищали с использованием микроколонок с SephadexТМ G-50 medium и секвенировали по Сэнгеру с использованием BigDye Terminator v3.1 Cycle Sequencing Kit и полимера POP-7 (Applied Biosystems, США) на генетическом анализаторе ABI 3500 (Applied Biosystems, США). Полученные хроматограммы были проанализированы с применением программного обеспечения Chromas. Все мутации были обозначены в соответствии с нумерацией нуклеотидов в первичном транскрипте микроРНК.
Предсказание insilico генов-мишеней для каждой из «ключевых» последовательностей микроРНК было выполнено с помощью специального онлайн-инструмента miRDB [22]. Для построения диаграмм Венна был использован электронный ресурс https://www.semestr.online/graph/venn.php#vennhand [23]. Для определения функции генов-мишеней микроРНК был проведен анализ обогащения терминов генных онтологий (GOenrichmentAnalysis) молекулярных функций и биологических процессов с помощью PANTHER [24] на базе биоинформационного ресурса Gene Ontology (GO; http://www.geneontology.org) [25]. Результаты со значением p<0,05 после поправки на множественную проверку гипотез (FDR – False Discovery Rate) считались статистически значимыми. Для предсказания вторичной структуры «шпильки» микроРНК и расчета ее минимальной свободной энергии (dG) использовался UNAfold web server [26].
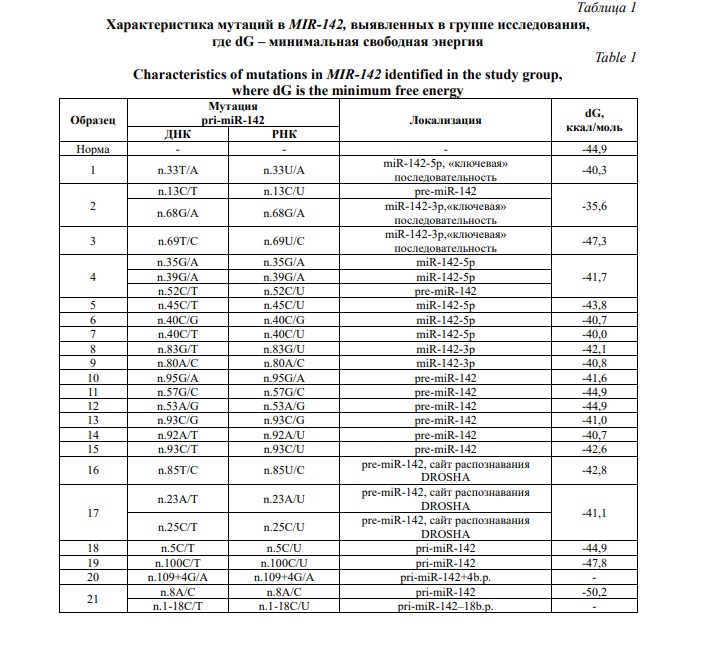
Результаты исследования. В исследованной группе образцов опухолевой ткани пациентов с ДВККЛ в 21/125 (16,8%) случаев были выявлены однонуклеотидные замены в MIR-142 в гетерозиготном состоянии. Всего 26 вариантов нуклеотидной последовательности (ВНП) (Табл. 1). Отметим, что в 3-х случаях имели место одновременно две находки (n.13C/T в сочетании с n.68G/A, а также n.23A/T в сочетании с n.25C/T и n.8А/С в сочетании с n.1-18C/T, в 1-м случае – три мутации в MIR-142 (n.35G/A, n.39G/A и n.52C/T) в образце (Рис. 1).
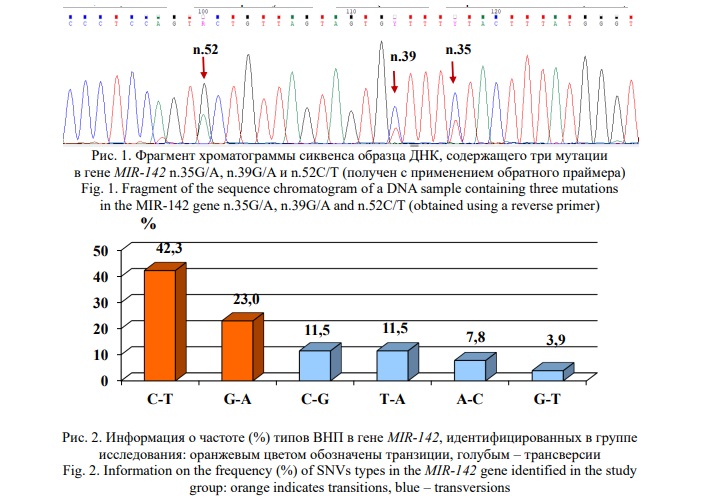
Анализ типов замен показал, что наиболее распространенными были транзиции между C и T (42,3%), а также G и A (23,0%). Среди трансверсий замены между C и G, а также Т и А составили по 11,5%, еще 7,8% составили замены между А и С и 3,9% - между G и Т.
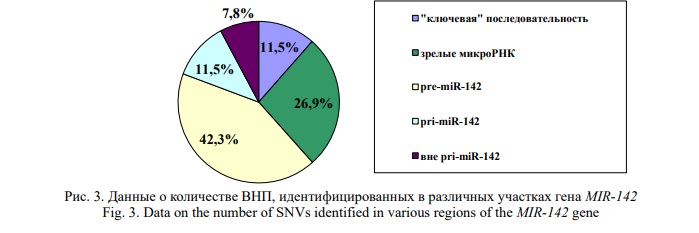
Анализ распределения ВНП по последовательности гена показал, что только 3-и из них (11,5%) были локализованы в «ключевых» последовательностях miR-142-5p и miR-142-3p (1 и 2 случая, соответственно), тогда как 7 (26,9%) – в других участках последовательности зрелых микроРНК вне сайтов связывания с мРНК-мишенями (4 и 2 случая, соответственно). Одиннадцать (42,3%) ВНП были локализованы в последовательности pre-miR-142 вне участков, кодирующих зрелые цепи, в том числе 3-и – в сайте распознавания DROSHA. Еще три (11,5%) ВНП – в последовательности pri-miR-142 за пределами участков, кодирующих микроРНК-предшественника. В двух случаях (7,8%), выявлены однонуклеотидные замены в непосредственной близости к 5/- и 3/-концам pri-miR-142 (Рис. 3).
Учитывая, что три из выявленных ВНП затрагивали «ключевые» последовательности зрелых цепей микроРНК, была проведена идентификация наборов генов-мишеней с помощью сервиса miRDB [22]. Прогнозирование проводилось для «ключевых» последовательностей miR-142-5р и miR-142-3р в норме и для каждого из мутантных вариантов.
Анализ выявил значительное изменение профиля предсказанных взаимодействий микроРНК-мишень (Рис. 4). В случае n.33U/A ключевой последовательности miR-142-5p имело место увеличение (с 1133 до 1328), тогда как в случае n.68G/A и n.69U/C ключевой последовательности miR-142-3p – уменьшение (с 418 до 312 и с 418 до 406, соответственно) числа потенциальных мишеней. При этом наблюдалось значительное изменение спектра взаимодействия, особенно выраженное для n.68G/A miR-142-3p. Согласно биоинформатическому предсказанию, при данной мутации сохраняется контроль лишь над 18/418 (4,3%) канонических мРНК мишеней, что значительно меньше, чем при n.69U/C miR-142-3p (145/418, 34,7%) и n.33U/A miR-142-5p (456/1133, 40,2%).

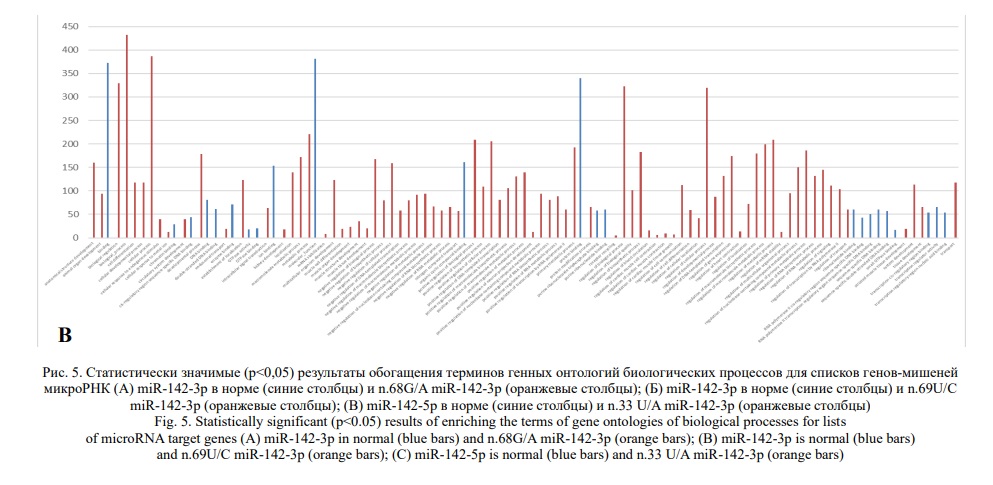
каждого набора генов-мишеней (референсного и измененного в результате каждой из мутаций). Анализ выявил статистически значимое (p<0,05) обогащение различных наборов терминов генных онтологий для miR-142-3p в норме и при n.68G/A или n.69U/C, а также для miR-142-5p в норме и при n.33U/A (Рис. 5).
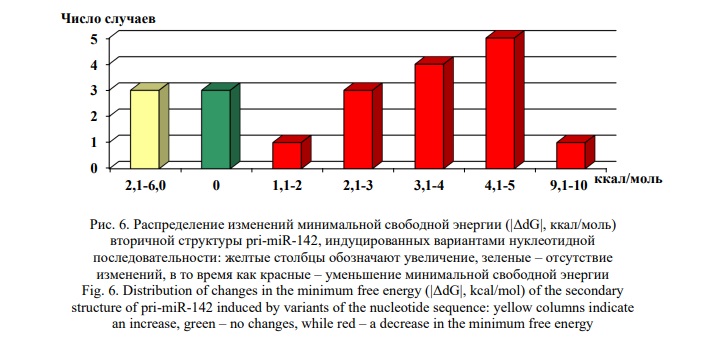
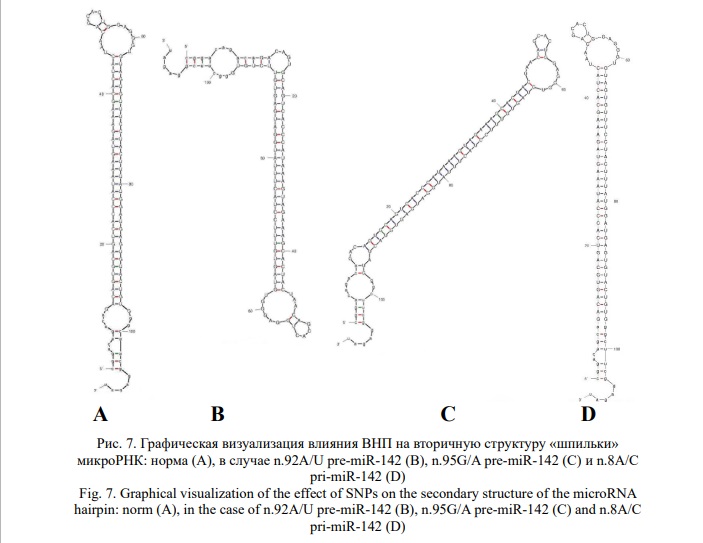
Чтобы оценить влияние выявленных замен на вторичную структуру «шпильки» и ее термодинамическую стабильность, использовался UNAfold web server [26]. Сначала была проанализирована вторичная структура pri-miR-142 в норме, далее каждого из 19 мутантных вариантов ее последовательности. Показано, что в 17/19 (89,5%) случаев ВНП приводили к ненулевому изменению минимальной свободной энергии (|ΔdG|) в диапазоне от 1,1 до 9,3 ккал/моль (Рис. 6). Только три из них приводили к приросту минимальной свободной энергии, а, следовательно, повышению термодинамической стабильности «шпильки», тогда как остальные – к их снижению. Более того, практически все ВНП влияли на структуру «шпильки». Примеры представлены на рисунке 7.
Обсуждение. Мутации в гене MIR-142 описаны в единичных случаях фолликулярной лимфомы [27, 28, 29], хронического лимфоидного лейкоза [30], а также острого миелоидного лейкоза и миелодиспластического синдрома [31, 32], но наиболее часто выявляются при ДВККЛ [33]. В данном исследовании подробно охарактеризована частота и спектр ВНП в гене MIR-142 в опухолевой лимфоидной ткани крупной выборки пациентов с ДВККЛ (123 человека) и провели анализ in silico их функционального эффекта.
Частота мутаций в группе исследования составила 16,8%, что укладывается в описанный в литературе диапазон – от 12 до 20% [27, 34, 35, 36], при этом в четырех случаях имели место множественные однонуклеотидные замены в последовательности MIR-142, что косвенно свидетельствует о возможности феномена катаэгиса при ДВККЛ в области расположения данного гена [37]. Под катаэгисом понимают явление гипермутаций, наблюдающееся при ряде злокачественных новообразований и характеризующееся локализованными кластерами однонуклеотидных замен – шесть или более в пределах тысячи п.н. [38].
Всего было выявлено 26 типов однонуклеотидных замен, широко разбросанных по последовательности гена, что отличает ДВККЛ от других типов гемобластозов. Так, согласно данным литературы, все известные в настоящее время ВНП в данном гене при остром миелоидном лейкозе и миелодиспластическом синдроме описаны только в «ключевой» области зрелой цепи miR-142-3p, ответственной за связывание генов-мишеней [31, 32, 39-41], как и единственная описанная в настоящее время мутация в MIR-142 при лимфоме Беркитта [18]. При фолликулярной лимфоме верифицированные ВНП затрагивают последовательность miR-142-3p [27, 28], а при хроническом лимфолейкозе – в большинстве случаев локализованы в miR-142-5p [21, 29, 30, 39].
Полученные данные позволяют утверждать, что в опухолевой ткани пациентов с ДВККЛ в гене MIR-142 транзиции преобладают над трансверсиями. Среди ВНП транзиции пиримидиновых оснований С-Т составили 42,3%, далее по частоте следовали транзиции пуриновых оснований А-G (23,0%). Трансверсии (замена пиримидинового основания на пуриновое, либо наоборот) суммарно составили лишь треть (34,7%) находок, что согласуется с данными по спектру однонуклеотидных замен в других генах при новообразованиях человека [42, 43]. Причинами данного явления считают, как более частое возникновение транзиций при работе ДНК-полимераз в клетках, так и их селекцию в ходе опухолевой прогрессии [44].
Анализ распределения ВНП в гене MIR-142 показал, что лишь небольшое их количество (11,5%) было локализовано в «ключевых» областях, большая же часть – за ее пределами: в последовательности зрелых цепей (26,9%), предшественника (42,3%) или первичного транскрипта (11,5%) микроРНК, что аналогично данным исследований других генов микроРНК. В двух случаях (7,8%), выявлены однонуклеотидные замены в непосредственной близости к 5/- и 3/-концам pri-miR-142. Наблюдающееся увеличение частоты ВНП в генах, кодирующих микроРНК, в направлении от «ключевой» области и зрелых последовательностей к последовательности микроРНК-предшественника, как полагают, связано с явлениями отбора в опухоли функционально-значимых замен [20]. Полученные данные могут свидетельствовать о том, что предшественник микроРНК-шпильки имеет довольно «хрупкую» структуру, и самые различные ВНП способны нарушать реализацию функции кодируемой микроРНК за счет изменения трехмерной организации предшественника.
В этой связи нами выполнялся биоинфортический анализ эффекта идентифицированных однонуклеотидных замен на переключение генов-мишеней, а также стабильность и вторичную структуру «шпильки» микроРНК. По данным базы miRDB канонический вариант miR-142-3р потенциально нацелен на 418, тогда как miR-142-5р – на 1133 мРНК-мишеней [22]. Анализ in silico влияния ВНП, расположенных в «ключевой» последовательности, на взаимодействия микроРНК-мРНК показал, что все три выявленные в группе исследования замены приводят к существенному изменению набора регулируемых генов. В случае n.33U/A 456 (40,2%) генов-мишеней оставались общими с канонической зрелой miR-142-5p, в то время как 677 (59,8%) были утрачены, а еще 872 приобретены. Аналогичная тенденции наблюдалась для n.69U/C: 145 (34,7%) общих с miR-142-3p в норме, утрата 273 (65,3%) и приобретение 161 новых генов-мишеней. Наиболее же выраженные изменения спектра регулируемых генов установлены для n.68G/A, при которой, согласно предсказанию сервиса miRDB, сохраняется контроль лишь над 18 (4,3%) каноническими мРНК мишенями miR-142-3p, тогда как 273 (65,7%) и 161 мишень утрачиваются и приобретаются, соответственно.
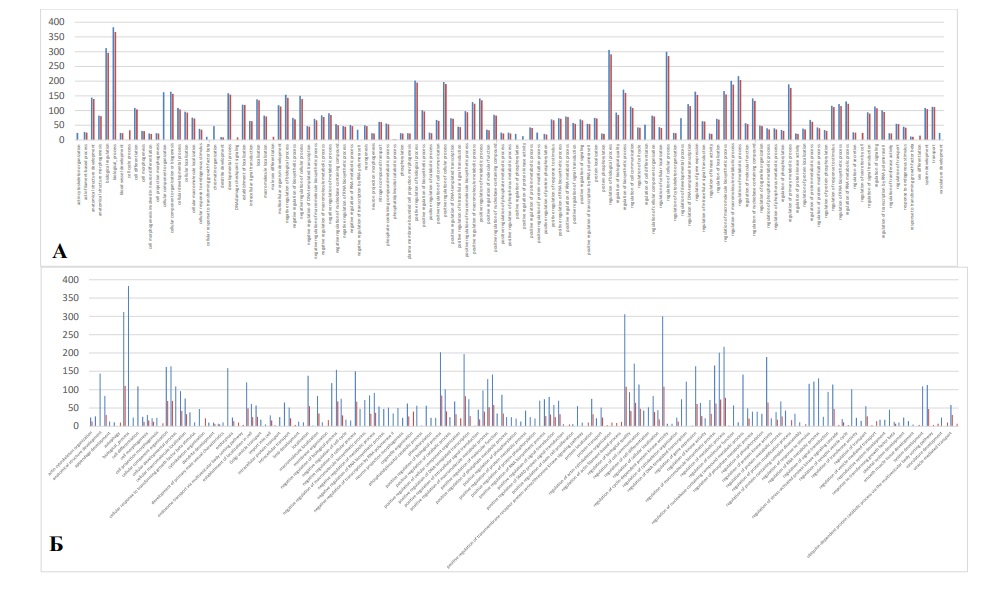
Чтобы выяснить, могут ли такие изменения в спектре генов-мишеней иметь дальнейшие последствия, влиять на биологические пути и, как следствие, на функционирование клеток, был проведен анализ обогащения терминов генных онтологий для полученных списков генов. Показано, что изменения в наборах генов-мишеней микроРНК в результате выявленных в группе исследования однонуклеотидных замен в областях «ключевых» последовательностей зрелых цепей miR-142 нашли свое отражение в генных онтологиях биологических процессов. Для всех трех мутаций идентифицированы отличные число и наборы терминов генных онтологий по сравнению с рефференсными микроРНК. Общая тенденция заключается в следующем. Несмотря на то, что для ВНП «ключевой» области miR-142-3p многие термины генных онтологий были общими с референсной последовательностью, в случае мутации количество задействованных генов-мишеней в одном и том же процессе для n.68G/A было несколько ниже, а для n.69U/C существенно ниже по сравнению с соответствующей нормальной зрелой цепью микроРНК. Важно отметить, что недавнее функциональное исследование n.69U/C и n.68G/A показало, что, несмотря на локализацию в miR-142-3p,обе они приводят к снижению уровня как miR-142-3p, так и miR-142-5p [18].
Среди факторов, на которые влияла n.68G/A, были жизненно важные клеточные процессы, связанные с клеточным ответом на стимул трансформирующего фактора роста β, организацией хроматина, регуляцией транскрипции РНК-полимеразой II, активности протеинкиназ, дифференцировки клеток, клеточного цикла, контрольной точки повреждения ДНК, а также развитие сосудистой сети.
В случае n.69U/C наиболее значимыми были генные онтологии, связанные с дифференцировкой клеток, клеточным ответом на стимул трансформирующего фактора роста-бета, организацией хроматина, внутриклеточной сигнальной трансдукцией, регуляцией метаболизма, клеточного цикла, транскрипции, процесса биосинтеза ДНК, макромолекул и соединений, аутофагии, трансмембранной передачи сигнала, убиквитин-зависимого катаболизма белков, системным развитием сосудистой сети, распространением клеток, зависящим от адгезии.
Тогда как для n.33U/A «ключевой» области miR-142-5p в сравнении с референсной последовательностью наблюдалось кардинальное изменение картины обогащения терминов генных онтологий биологических процессов регуляции дифференцировки клеток, клеточного ответа на эндогенный стимул, внутриклеточной передачи сигналов, транскрипции, стабильности мРНК, биосинтеза и метаболизма макромолекул и клеточного роста.
Вместе с тем конкретный перечень генов, регулируемых каждым из мутантных вариантов, являются предсказанными и, безусловно, нуждаются в дальнейшей экспериментальной проверке. Так, например, описаны случаи, когда изменения в «ключевой» последовательности микроРНК не приводили к полной утрате контроля над мишенью: даже в случае неточных взаимодействий сохранялась та или иная степень репрессивной активности [45]. С другой стороны, установлена возможность связывания микроРНК с 5/-НТП мРНК или взаимодействия между 3/-НТП мРНК и микроРНК вне «ключевой» последовательности, что не учитывается в ходе анализа insilico [46].
Следует отметить также, что мутации во всех трех положениях гена MIR-142 были описаны ранее при гемобластозах [39]. ВНП в положении n.69 по данным работ других авторов являются наиболее частыми, в основном представлены заменой U/C и зарегистрированы при ДВККЛ, фолликулярной лимфоме и хроническом лимфоидном лейкозе. Замена n.68G/A также уже регистрировалась при ДВККЛ, тогда как при ОМЛ ВНП в данном положении были представлены другой заменой – G/С. В позиции n.33 ранее при фолликулярной лимфоме была описана отличная от выявленной нами замены U/A замена U/С.
Большинство ВНП, идентифицированных в данном исследовании, расположены за пределами «ключевой» области, следовательно, они не оказывают прямого влияния на выбор гена-мишени микроРНК. В литературе имеются данные о том, что ВНП, расположенные в последовательности предшественника микроРНК, оказывают существенное влияние функционирование микроРНК, начиная с ядерного и цитоплазматического этапов созревания и заканчивая термодинамической стабильностью, формированием вторичной структуры, отбором цепей, включаемых в RISC, что в конечном итоге отражается на уровне их экспрессии [47].
Три из ВНП, выявленных нами, затрагивали сайт распознавания DROSHA. Их прямым функциональным эффектом может быть нарушение созревания pri-miR-142 до pre-miR-142. Однако, все без исключения выявленные замены потенциально могут влиять на вторичную структуру предшественников микроРНК и оказывать существенное влияние на биогенез зрелых цепей [45].
С применением UNAfold web server нами были определены наиболее стабильная (в термодинамическом равновесии) вторичная конформация «шпильки» микроРНК и ее минимальная свободная энергия (dG), которые зависят от таких факторов как длина молекулы, состав нуклеотидов, а также последовательность их расположения в цепочке. Минимальная свободная энергия может быть рассчитана путем сложения свободных энергий компонентов: взаимодействующих комплементарно пар оснований, выпуклостей и петель. При этом в ряду микроРНК одинакового размера наиболее структурированной и стабильной считается та, которая имеет наибольшее отрицательное значение минимальной свободной энергии [48].
Анализ показал, что в 89,5% случаев, выявленные в группе исследования ВНП приводили к изменению минимальной свободной энергии в диапазоне от 1,1 до 9,3 ккал/моль. Практически все они приводили к снижению минимальной свободной энергии, а, следовательно, уменьшению стабильности «шпильки». Еще три не сопровождались какими-либо сдвигами и, возможно, не влияют на стабильность микроРНК. Тогда как еще три замены (упомянутая ранее n.69T/C, а также n.8A/C и n.100C/T), согласно предсказанию UNAfold web server, могут стабилизировать вторичную структуру молекулы. Стоит отметить, что Gong J. et al. обобщили имеющиеся исследования в этой области и установили, что изменения минимальной свободной энергии более, чем на 2,0 ккал/моль существенно влияют на выработку зрелой микроРНК [49], но даже изменения ниже этого порога могут изменять их биогенез [50]. Также практически все описанные нами ВНП, за исключением трех, в той или иной степени, иногда существенно влияли на структуру микроРНК «шпильки».
Заключение. Частота мутаций в гене MIR-142 в группе исследования составила 16,8%. При этом в каждом пятом случае имели место множественные однонуклеотидные замены, что косвенно свидетельствует о возможности феномена катаэгиса при ДВККЛ в области расположения данного гена. В целом полученные результаты свидетельствуют о существенном влияние ВПН в «ключевых» последовательностях на переключение генов-мишеней зрелых цепей изучаемой микроРНК, тогда как самые различные ВНП способны нарушать трехмерную организацию «шпильки» микроРНК предшественника. Необходимы дальнейшие функциональные исследования для подтверждения влияния предсказанных изменений термодинамической стабильности и вторичной структуры на биогенез зрелых цепей микроРНК.
Информация о финансировании
Работа выполнена за счет средств гранта Российского Научного Фонда № 25-25-00068.





















Список литературы