Роль транспозонов в обеспечении эволюционного разнообразия протеомов эукариот (обзор)
Aннотация
Актуальность: Транспозоны составляют значительную долю геномов эукариот и являются объектами для современных генетических исследований, в том числе в проектировании таргетной терапии опухолей. Для оптимального проектирования таких методов важно определить возможные взаимосвязи мобильных генетических элементов с регуляторными элементами генома в связи с происхождением от транспозонов многих белок-кодирующих генов и способных к трансляции некодирующих РНК. Цель исследования:Описать роль мобильных генетических элементов в обеспечении эволюционного разнообразия протеомов эукариот как за счет непосредственного возникновения белок-кодирующих генов от транспозонов, так и путем возникновения от них некодирующих РНК, способных к трансляции. Определить практическую ценность полученных результатов путем анализа вовлечения образуемых из некодирующих РНК пептидов в канцерогенезе. Материалы и методы:Использованы базы данных Scopus, WoS, PubMed для анализа роли транспозонов в возникновении белок-кодирующих генов, микроРНК, длинных некодирующих РНК и кольцевых РНК, об участии образуемых при трансляции данных молекул РНК пептидов в канцерогенезе. Результаты:Согласно проанализированной литературе, транспозоны являются важнейшими источниками возникновения и эволюции белок-кодирующих генов за счет одомашнивания генов мобильных генетических элементов, экзонизации инсертированных ретроэлементов и образования псевдогенов. Большинство длинных некодирующих РНК, кольцевых РНК, многих генов микроРНК, а также их регуляторных элементов в эволюции произошли от транспозонов. У человека достоверно выявлена трансляция 15 длинных некодирующих РНК, 4 при-микроРНК и 6 кольцевых РНК с образованием функциональных пептидов, вовлеченных в механизмы канцерогенеза. Заключение:Возникновение в эволюции большинства генов некодирующих РНК и многих белок-кодирующих генов от транспозонов свидетельствует о возможности использования данных геномных элементов в качестве мишеней для перспективных генетических исследований, в том числе в лечении заболеваний у человека. Свидетельством служат полученные данные о вовлечении в канцерогенез 25 пептидов, образуемых при трансляции некодирующих РНК
Ключевые слова: белок-кодирующие гены, канцерогенез, длинные некодирующие РНК, микроРНК, транспозоны, эволюция
Введение. Транспозоны или мобильные генетические элементы (МГЭ) представляют собой специфические локусы генома, способные к перемещениям в новый локус того же генома с помощью ферментов, кодируемых их собственными генами (автономные МГЭ) или генами других МГЭ (неавтономные). У эукариот различают класс I МГЭ – ретроэлементы (РЭ) и класс II – ДНК-транспозоны. МГЭ класса I перемещаются за счет обратной транскрипции собственных РНК со встраиванием кДНК в новый локус генома по механизму «копирования и вставки». Их классифицируют на порядки LTR, DIRS, PLE, LINE, SINE. ДНК-транспозоны классифицируют на подкласс 1, включающий TIR и Crypton и подкласс 2, содержащий порядки Helitron и Maverick [1]. Геномы большинства бактерий и археев практически не содержат МГЭ, за некоторыми исключениями. Например, у бактерии Clostridiumdifficile МГЭ составляют 11% последовательностей генома, у Enterococcusfaecalis – 25%, у Orientiatsutsugamushi – 46,7% и отличаются способностью взаимодействовать между собой путем рекомбинации, формируя новые химерные элементы [2]. Однако наибольшую регуляторную и эволюционную роль МГЭ приобрели у эукариот, о чем свидетельствует их глобальная распространенность и использование последовательностей МГЭ для образования и эволюции новых белок-кодирующих генов (БКГ) и некодирующих РНК (нкРНК). Благодаря этому эукариоты характеризуются выраженными различиями в размерах геномов. Например, у растений наиболее мелкий геном микроспоридии Encephalitozoonintestinalis размером в 2,3 миллиард п.н. меньше крупного генома Parisjaponica (148 852 милиард п.н.) в 70 000 раз, а количество нуклеотидов ДНК амебы почти в 200 раз больше по сравнению с геномом человеком [3], который представляет наибольший интерес в современных исследованиях в связи с открывающимися возможностями геномного редактирования.
В ядерной ДНК человека LINE занимают 0,63 млрд. п.н., SINE – 0,39 млрд. п.н., LTR – 0,27 млрд. п.н., ДНК-транспозоны – 0,108 млрд. п.н. Всего МГЭ составляют около 1,4 млрд. п.н., что составляет 46,7% всех последовательностей генома [4]. При помощи специфических олигонуклеотидов, комплементарных транспозонам, проведенный анализ генома человека показал, что последовательности МГЭ занимают не менее 2/3 всего генома человека [5]. Такое несоответствие обусловлено ключевой ролью МГЭ в возникновения и эволюции генов нкРНК и белок-кодирующих генов, что предполагает многофункциональность таких генов как специфическое свойство МГЭ [6], поскольку при возникновении мира ДНК-РНК-белки из мира РНК-ДНК, транспозоны служили универсальными источниками происхождения бесчисленного разнообразия пептидных и белковых молекул благодаря процессингу их транскриптов [7]. Соответственно, транскрипты МГЭ процессируются различными ферментативными системами с образованием множества вариантов молекул РНК, которые могут связываться с рибосомами и формировать огромное разнообразие белков и пептидов. Действительно, в эволюции МГЭ становились источниками не только множества БКГ [8-19], но также генов микроРНК [20] и длинных некодирующих РНК (днРНК) [21, 22].
Согласно опубликованной в 2022 году статье о расшифровке полной последовательности генома человека, в гаплоидном наборе содержится 3,055 миллиардов пар нуклеотидов. Всего выявлено 63494 генов, из которых лишь 19969 БКГ [4]. Значительную долю составляют гены нкРНК, количество которых в базе данных GENECODE все более возрастает благодаря современным молекулярно-генетическим методам исследования. В 2024 году статистические данные этой базы (https://www.gencodegenes.org/human/stats.html) свидетельствуют о наличии 20424 генов днРНК, ответственных за образование 59719 транскриптов. Аннотировано 7565 генов малых нкРНК, а также 14719 псевдогенов [4], которые также образованы в эволюции благодаря МГЭ [23, 24]. Происхождение генов нкРНК [20, 21, 22] и БКГ [8-19] от МГЭ предполагает наличие специфических свойств таких генов и продуктов транскрипции, отражающих универсальные свойства МГЭ. Анализ научной литературы показывает появление сведений, подтверждающих это предположение. И в данной статье будут представлены доказательства того, что нкРНК являются важнейшими источниками возникновения новых белок-кодирующих генов в эволюции, что связано с их функциональной взаимосвязью с МГЭ. Исследование роли МГЭ в обеспечении разнообразия транскриптомов и протеомов имеет практическое значение, поскольку транспозоны являются мишенями для проведения таргетной терапии злокачественных новообразований [25]. Соответственно, полученные результаты о значении МГЭ в происхождении белков и нкРНК могут свидетельствовать о возможном вовлечении этих молекул в канцерогенез.
Цель исследования. Описать роль МГЭ в обеспечении эволюционного разнообразия протеомов эукариот как за счет непосредственного возникновения белок-кодирующих генов от МГЭ, так и путем возникновения от транспозонов некодирующих РНК, способных к трансляции. Определить практическую ценность полученных результатов путем анализа вовлечения образуемых из нкРНК пептидов в канцерогенезе.
Материалы и методы исследования. Использованы базы данных Scopus, WoS, PubMed для анализа роли транспозонов в возникновении белок-кодирующих генов, микроРНК, длинных некодирующих РНК и кольцевых РНК, об участии образуемых при трансляции данных молекул РНК пептидов в канцерогенезе.
Результаты. Анализ научной литературы позволил выявить важную роль МГЭ в возникновении БКГ и различных нкРНК. Возможность трансляции таких нкРНК обеспечивает образование функциональных пептидов, многие из которых участвуют в регуляции канцерогенеза и могут быть использованы в качестве инструментов для таргетной противоопухолевой терапии.
Роль транспозонов в возникновении белок-кодирующих генов
Формирование в эволюции новых БКГ возможно тремя отдельными механизмами при помощи МГЭ: 1) путем непосредственного одомашнивания генов МГЭ, 2) за счет экзонизации инсертированных в интроны или некодирующие области существующих БКГ последовательностей МГЭ (с образованием новых вариантов сплайсинга) и 3) путем образования ретрогенов (псевдогенов) в результате использования ферментов РЭ (рис. 1). Даже формирование псевдогенов способствует изменению генных регуляторных сетей, так как ретрокопии фланкированы последовательностями МГЭ [26], находящимися под управлением сложных эпигенетических систем, с участием происходящих от МГЭ некодирующих РНК [6]. Cплайсосомные интроны также произошли от МГЭ в эволюции [24], с последующими новыми инсерциями МГЭ в эти интроны, что способствовало формированию альтернативных сплайсинговых транскриптов. Если в ходе эволюции образованные новые белковые изоформы способствовали лучшей адаптации, эти варианты закреплялись на уровне вида. Возникала экзонизация. В геноме человека выявлены различные экзоны, произошедшие от МГЭ [27]. С помощью модели глубокого обучения eXAlu, было показано, что количество элементов Alu в геноме человека, подверженных экзонизации составляет около 110 000, что в 21 раз больше, чем представлено в базе данных GENOCODE [28].
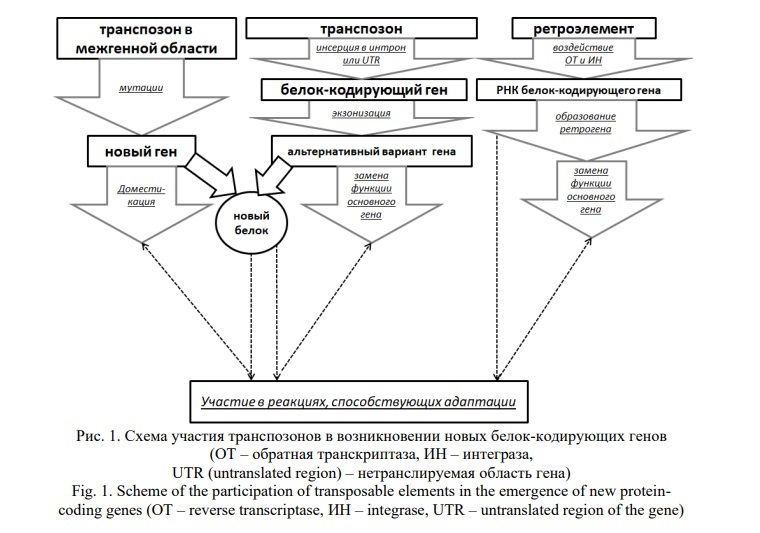
При образовании ретрогенов используются кодируемые ретроэлементами обратная транскриптаза для образования кДНК из транскриптов БКГ и эндонуклеаза для встраивания копии в геном. В результате образованные ретрогены содержат новые регуляторные последовательности, содержащиеся в локусе встраивания. Кроме того, такие новые БКГ во фланкирующих областях содержат специфические для LINE последовательности, такие как удвоенные целевые сайты и последовательности расщепления эндонуклеазы ТТТТ/АА. У животных описано также участие ферментов ERV (эндогенных ретровирусов) в образовании ретрогенов. В таких случаях новые БКГ будут фланкированы длинными концевыми повторами (LTR), характерными для ERV [23]. Однако использование LTR-содержащих РЭ при образовании псевдогенов наиболее характерно для растений, поскольку в их геномах, по сравнению с животными, преобладают LTR-содержащие РЭ. Например, у арабидопсиса описаны фланкированные длинными концевыми повторами функциональные ретрогены CYP98A8 и CYP98A9, а у томатов – псевдоген Sun [29]. У растений отмечена также способность ретрогенов транскрибироваться на более низком уровне (подобно генам днРНК), что отражает свойства происходящих от МГЭ генов днРНК в эволюции. Например, у OryzaL. более 2/3 всех псевдогенов экспрессируются со специфическими для тканей особенностями, что также отражает сходство с генами днРНК [30].
Подобно генам днРНК, эволюционно молодые псевдогены, состоящие только из экзонов, постепенно приобретают мозаичную структуру за счет многократных встраиваний новых МГЭ, которые содержат донорные и акцепторные сайты сплайсинга, обеспечивая взаимодействие мРНК таких генов со сплайсосомой. Это дает возможность участвовать белковым продуктам таких генов в регуляторных сетях, в которых задействованы МГЭ. Соответственно, такие псевдогены в определенных условиях обеспечивают лучшую адаптацию особей и могут быть отобраны для экспрессии на более высоком уровне и использоваться вместо БКГ, копией которого они являются [24]. Кроме того, образованные ретрокопии служат источниками транскрипции некодирующих РНК, что было доказано при анализе распространения кластеров микроРНК с участием Alu и L1 в геноме человека. За счет этого образуются новые возможности взаиморегуляции с другими генами и локусами генома, что потенциально служит источником новых адаптивных функций [31].
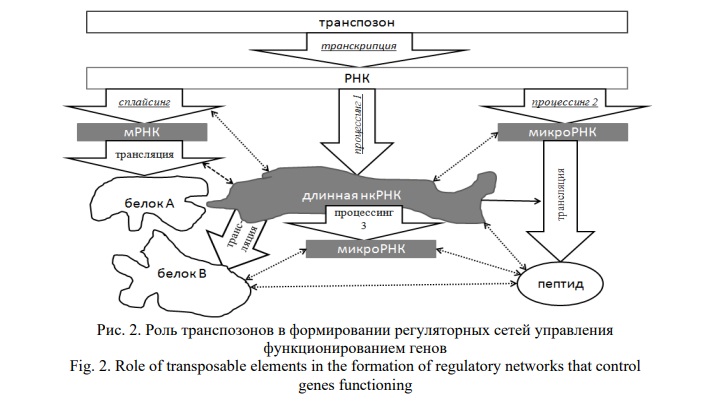
Для определения происхождения БКГ от транспозонов используют различные подходы, одним из которых является идентификация консервативных доменов, специфичных для МГЭ. В результате можно выявить множество генов, возникших в эволюции из последовательностей МГЭ и используемых для нужд хозяев. Некоторые из таких БКГ образуют тандемные кластеры семейств генов [32]. Формирование новых генов путем вербовки доменов МГЭ является причиной их распространения в связи с возможностью участия в адаптивных реакциях. Доказательства одомашнивания МГЭ в эволюции были получены в связи с наличием функциональных различий между автономными и доместицированными транспозонами [15]. МГЭ оказались источниками возникновения консервативных генов, играющих ключевую роль в эволюционных преобразованиях живых организмов. Например, обратная транскриптаза РЭ стала основой для формирования теломеразы [27]. У всех эукариот было идентифицировано множество БКГ, произошедших от МГЭ [10]. Так, у позвоночных описано более 1000 генов, источниками которых оказались РЭ [33]. Древняя эволюционная способность МГЭ образовывать белковые структуры, взаимодействующие с последовательностями собственных ДНК в геноме стала основой для возникновения от МГЭ различных транскрипционных факторов и сайтов связывания с ними, формируя сложные регуляторные генные сети [10, 34], находящиеся также под регуляторным влиянием МГЭ и произошедших от них некодирующих РНК (рис. 2).
Несмотря на то, что в геномах эукариот преобладают РЭ, важным источником БКГ различных животных, грибов и растений стал ген транспозазы ДНК-транспозонов (табл. 1). От транспозазы произошли такие консервативные белки как центромер-связывающий протеин CENP-B у животных, белок harbi1 у млекопитающих, рыб и лягушек. Важнейшие компоненты иммунной системы RAG у позвоночных также произошли от транспозазы, которая стала также источником возникновения белков Metnase и Pgbd у человека и мыши, протеинов BUSTER1-3, ZBED1, ZBED4, ZBED5, P52rlPK у млекопитающих. У арабидопсиса от транспозазы произошел белок Daysleeper [9, 15]. Специфические нуклеазы, используемые для перегруппировки ДНК у животных, произошли от транспозазы ДНК-транспозонов Harbinger [8]. Транспозазы стали основой для формирования таких ДНК-связывающих доменов эукариот как спираль-поворот-спираль (НТН) и цинковые пальцы (ZF). Кроме того, от транспозазы произошли HD (гомеодомен), KRAB (Kruppel-связанный бокс), BTB (Broad-Complex, Tramtrack, and Bric-a-brac), SET (Su(var), E(z) and Trithorax), SWIM (SWI2/SNF2 and MuDR), hATC (hAT C-terminal dimerization), LZ (лейциновая молния) [10]. Следует отметить роль ДНК-транспозонов в качестве эволюционных источников генов белков, участвующих в эпигенетической регуляции. Так, от ДНК-транспозона Harbinger у Арабидопсиса произошли гены HDP1 (от транспозазы) и HDP2 (от ДНК-связывающего белка), которые взаимодействуют с компонентами IDM1, IDM2, IDM3 и MBD7 ацетилтрансферазного комплекса, участвующего в деметилировании ДНК [18]. От транспозазы hAT произошли факторы модификации хроматина BEAF-32 и HIM-17 [11], центромерный белок Abp1 (у грибов) [10], инсуляторы, участвующие в модификации хроматина. Инсуляторы – это регуляторные элементы, которые способствуют организации хроматина эукариот за счет блокирования энхансеров и активации хроматинового барьера [17]. Гены, произошедшие от ДНК-транспозонов, экспрессируются в белки THAP0, THAP1, E93 (ген Eip93F), участвующие в апоптозе, а также THAP, LIN-36, LIN-15B, контролирующие клеточный цикл [11]. У дрожжей интеграза ДНК-транспозонов стала источником возникновения гена Fob1p, продукт которого управляет рекомбинацией рРНК [9, 15].
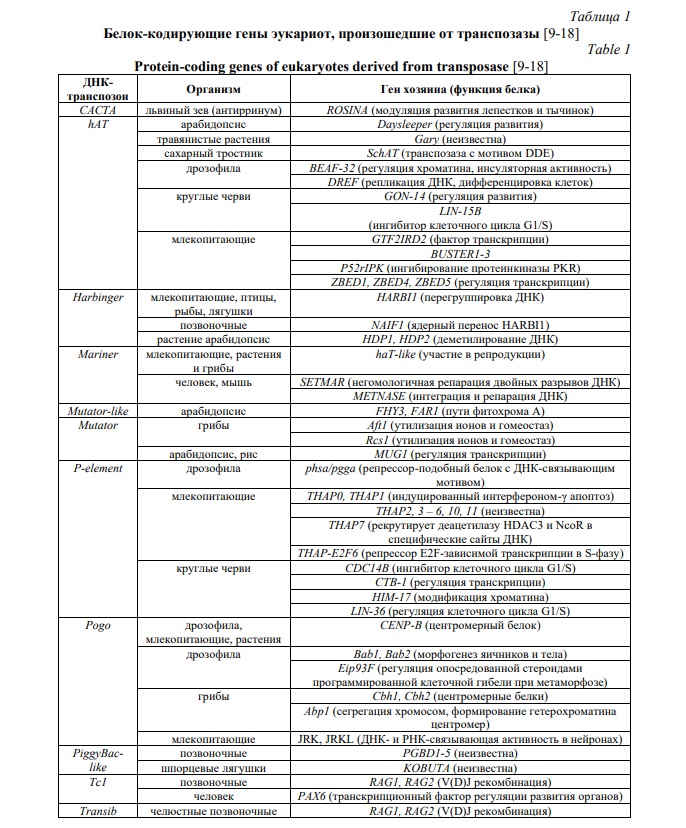
От гена интегразы LTR-ретроэлементов произошел ген Gin-1 у млекопитающих, белковый продукт которого участвует в регуляции эмбриогенеза. От генов GAG ретроэлементов произошли гены, вовлеченные в иммунные ответ (Ma), в управление транскрипцией миелина (MyEF-3), в регуляцию апоптоза и пролиферации клеток (Mart), противовирусный ответ (Fv1), регуляцию экспрессии отцовских генов (Rtl1), партеногенетическое развитие (PEG10) [9, 15]. Ранее предполагалось, что обратная транскриптаза и подобные ей ферменты относятся исключительно к РЭ или вирусам, а к происходящим от обратной транскриптазы генам относились лишь теломеразы. Однако было выявлено, что от гена обратной транскриптазы РЭ произошли гены rvt, которые содержатся в геномах бактерий, протистов, грибов, животных и растений с неоднородным филогенетическим распределением [13]. Обратная транскриптаза ERV эволюционировала в белок Prp8, являющийся компонентом сплайсосомы эукариот [12]. У млекопитающих в эволюции были одомашнены гены Env, кодирующие оболочку ERV, c образованием генов синцитинов Syncytin-1, -2, -A, -B, важной функцией которых является регуляция развития плаценты [9, 15].
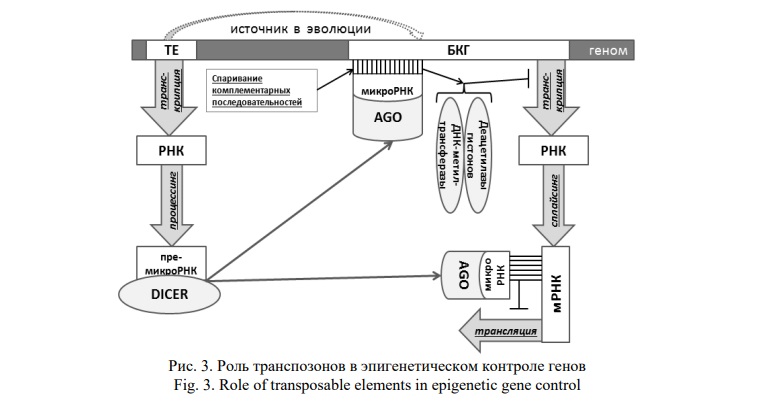
Белки, произошедшие от Env LTR-ретроэлементов, функционируют в противовирусной защите хозяев у позвоночных. Они образуют поверхностную (детерминирует клеточную специфичность, тропизм к хозяину и типу клеток) и трансмембранную (необходима для слияния оболочек вируса и клетки-мишени) субъединицы. Во всех случаях эндогенные env гены, произошедшие от генов различных ERVs, действуют как факторы ограничения для родственных экзогенных ретровирусов [16]. От ORF1 LINE-ретроэлементов у человека и других млекопитающих в эволюции произошел ген L1TD1, который кодирует РНК-связывающий белок, функционирующий в недифференцированных клетках [19]. Доместикация генов МГЭ для нужд хозяев приводит к тому, что новые БКГ содержат последовательности транспозонов не только в своих интронах и регуляторных областях, но и в консервативных экзонах. Это дает основу для эпигенетического контроля работы БКГ на транскрипционном и посттранскрипционном уровнях с участием происходящих от транспозонов нкРНК в качестве универсальных инструментов РНК интерференции (рис. 3).
Происхождение некодирующих РНК от транспозонов
К некодирующим РНК относятся днРНК длиной более 200 нуклеотидов и малые нкРНК (короче 200 нуклеотидов, включая микроРНК длиной 20-24 нуклеотида). МикроРНК негативно регулируют их целевые мРНК путем связывания с их 3’-нетранслируемой областью и подавляя трансляцию или нарушая стабильность молекул. Биогенез микроРНК состоит из процессинга более крупных молекул при-микроРНК в более короткие пре-микроРНК с дальнейшим их процессингом до зрелых микроРНК. Одна микроРНК может регулировать множество мишеней, участвуя в разнообразных функциях организма. Например, онкосупрессорная miR-34a ингибирует экспрессию 700 отдельных БКГ [35]. Гены днРНК могут располагаться как между БКГ, так и внутри них. Названия многих днРНК происходят в соответствии с БКГ, вблизи или в которых они расположены. Если они комплементарны генам, то называются антисмысловыми днРНК или антисмысловыми транскриптами (NAT – natural antisense transcript). Различают cis-NAT (перекрываются с комплементарными генам) и trans-NAT (транскрибируются от псевдогенов) [36]. Экспрессия генов днРНК имеет много общего с БКГ, поскольку днРНК транскрибируются РНК-полимеразой II, подвергаются сплайсингу и кэпированию [37].
Молекулы днРНК способны самостоятельно выполнять каталитические функции и называются рибозимами, однако в большинстве случаев они являются структурными компонентами рибонуклеопротеинов, играющих важные роли в клетках. Примерами являются рибосомы, представляющие собой сложные рибонуклеопротеидные комплексы. ДнРНК участвуют в регуляции транскрипции путем взаимодействуя с ДНК-связывающими белками, с гистон-модифицирующими комплексами и РНК-полимеразой, за счет чего служат своеобразными драйверами эпигенетической регуляции [38]. К таким днРНК относятся Airn, ecCEBP, H19, Kcnq1ot1, PAPAS, pRNA, PTENpa1-AS, TARID, Xist, которые вызывают образование метилцитозина в специфических локусах; ANRASSF1, ANRIL, BORDERLINE, Kcnq1ot1, NeST, PINT – являются гидами для факторов модификации гистонов. ДнРНК AK141205, AK028326, ES1, ES2, ES3, linc-RoR, Evx1as, Hoxb5/6as регулируют транскрипцию белок-кодирующих генов путем взаимодействий с факторами транскрипции. Кроме того, днРНК обладают сходными с микроРНК свойствами, влияя на экспрессию генов за счет ингибирования трансляции мРНК. К таким днРНК относятся Uchl1-as1, lincMD1, lincRNA-p21, ½-sbsRNA [37]. Некоторые днРНК (lincRNA-Cox2, lincRNA-p21) формируют рибонуклеопротеины, которые регулируют транскрипцию специфических наборов генов. Например, lincRNA-p21 вместе с hnRNP-K образуют комплексы, связывающиеся со специфическими областями генома и подавляющие транскрипцию генов по пути р53 [36].
ДнРНК могут выполнять функции приманки для транскрипционных факторов путем мимикрии последовательности или структуры ДНК-мишеней. К таким днРНК относятся gas5, PANDA, DHFRminor, Lethe. Общее происхождение днРНК и микроРНК от МГЭ предполагает наличие у них идентичных и комплементарных последовательностей, благодаря чему реализуется важная функция днРНК в качестве губок для микроРНК. Эта способность позволяет ингибировать специфические микроРНК [36]. Действительно, согласно литературным данным, последовательности многих зрелых микроРНК [20] и днРНК [21] идентичны или комплементарны последовательностям МГЭ. Большинство (более 80%) генов днРНК содержат один и более фрагмент МГЭ, а почти половина всех экзонов днРНК имеют транспозонное происхождение. Доместикация последовательностей МГЭ в эволюции обусловлена способностью РНК-структур МГЭ образовывать функциональные домены [22].
Одной из причин одомашнивания геномами хозяев генов МГЭ в качестве источников днРНК является функциональность транскриптов транспозонов [6], что связано с их древнейшими универсальными свойствами в качестве источников жизни на Земле [7]. Например, Alu встраиваются в гены днРНК и формируют структуры, необходимые для взаимодействия с мРНК за счет коротких несовершенных спариваний нуклеотидов. Регуляторными сигналами для днРНК обладают также LTR-содержащие РЭ. Например, в геноме человека выявлены тысячи генов днРНК, происходящие от LTR-РЭ [39], которые контролируют сеть плюрипотентности путем изменения структуры хроматина. Они участвуют в формировании бластоцисты и в дальнейшем эмбриогенезе [40]. Кроме того, LTR-РЭ могут непосредственно служить генами днРНК [41]. Транскрипты LINE1 также сами функционируют в качестве днРНК, взаимодействуя со специфическими участками хроматина и регулируя экспрессию генов в раннем эмбриогенезе. Например, при связывании с Nucleolin и KAP1, они вызывают как активацию генов рДНК, так и подавление многих генов двуклеточного эмбриона путем сайленсинга Dux [42]. То есть, помимо транспозиций с помощью белковых продуктов, подвергнутые альтернативному процессингу транскрипты РЭ способны функционировать самостоятельно в качестве молекул некодирующих РНК, что обусловлено их древним свойством, сформированном при возникновении жизни в мире РНК-ДНК [7]. Соответственно, взаимосвязи транспозонов с днРНК и их роль в возникновении новых БКГ могут отображать сходные процессы на ранних этапах эволюции живого, когда появилась преемственность функций рибозимов белковыми молекулами.
Тканевая специфичность днРНК превышает таковую для белков. При этом в регуляции дифференцировки стволовых клеток они взаимодействуют с МГЭ [43], что характерно для днРНК, образующихся из межгенных областей геномов эукариот, а также из перекрывающихся и антисмысловых паттернов относительно примыкающих БКГ, которые они и регулируют [44]. Это позволяет им в значительной степени детерминировать разнообразие клеточных фенотипов, особенно в нейронах центральной нервной системы [45]. Пространственные 3D-структуры молекулы днРНК характеризуются модульной организацией с образованием специфических доменов, которые состоят главным образом из последовательностей МГЭ [22]. Способность транскриптов МГЭ подвергаться процессингу с образованием мРНК или функциональных нкРНК с помощью рибонуклеаз, представляет собой эволюционный процесс приспособления генных сетей к меняющимся условиям при участии новых некодирующих РНК и их транскриптов [46, 47, 48]. В результате из наиболее оптимальных в естественном отборе генов нкРНК формируются новые БКГ [49-52]. Многофункциональность некодирующих РНК, происходящих от МГЭ, проявляется в том, что некоторые молекулы микроРНК могут образовываться из транскриптов днРНК [53]. Соответственно, такие днРНК являются также генами микроРНК, открытая рамка считывания прекурсоров которых может связываться с рибосомами с образованием функциональных белковых или пептидных молекул [54, 55, 56]. МГЭ также сами являются важными источниками генов микроРНК. Еще в 2016 году была опубликована база данных MDTE DB, в которую были включены 661 микроРНК человека, произошедшие от МГЭ [20].
Происхождение пептидов и белков от некодирующих РНК в эволюции
Возникновение днРНК и микроРНК от МГЭ в эволюции могло стать причиной многофункциональности данных нкРНК. Это означает, что, несмотря на название «некодирующие», они обладают грандиозным потенциалом трансляции в функциональные белки и пептиды. Причиной является универсальное свойство МГЭ содержать последовательности нуклеотидов, связывающихся с рибосомами даже при наличии неканонической короткой открытой рамки считывания (ORF – open reading frame), что обусловлено ролью МГЭ в качестве универсальных эволюционных источников жизни на Земле [7]. В нескольких филогенетических исследованиях было выявлено происхождение эволюционно новых БКГ различных эукариот от генов днРНК [49-52]. Так, у гриба Saccharomyces cerevisiae выявлен новый ген BSC4, содержащий ORF (которая отсутствует у других близкородственных видов грибов), кодирующую белок длиной 132 аминокислот. Продукт гена BSC4 участвует в путях восстановления ДНК в стационарную фазу гриба и способствует его устойчивости при перемещении в среду с низким содержанием питательных веществ. У грибов видов S. paradoxus, S. mikatae, S. bayanus идентичные некодирующие последовательности также транскрибируются, поэтому их можно отнести к категории гена днРНК, который у Saccharomyces cerevisiae в эволюции стал источником нового БКГ, доместицированного в связи с адаптивными функциями [50]. Сравнительный анализ генома мухи Drosophila melanogaster с другими видами рода Drosophila позволил выявить 5 генов, 4 из которых расположены на Х хромосоме и транслируются в пептиды длиной 58, 79, 97 и 158 аминокислот, 1 ген – на 2 хромосоме с кодированием пептида из 127 аминокислот. Данные гены произошли от некодирующих последовательностей ДНК, транскрибируемых у других видов (гены днРНК) [49].
В 2012 году были выявлены 24 эволюционно молодых БКГ, которые произошли в эволюции от генов днРНК. Из них 11 генов кодируют специфичные только для человека белки, а 13 других – консервативные для человека и шимпанзе. Эти гены содержат от 1 до 7 экзонов, а длина продуктов трансляции генов варьирует от 72 до 423 аминокислот [51]. Такие гены называют «орфанными», то есть характерными для специфического вида животного и не обнаруживаемые у других видов, что связано с их недавним возникновением в эволюции из генов днРНК и транспозонов и участием в видоспецифических адаптивных реакциях. Около 53% «орфанных» генов приматов содержат последовательности МГЭ, что свидетельствует об использовании их в качестве источников происхождения данных генов [57]. Подтверждение роли днРНК в качестве источников «орфанных» генов было получено в исследованиях 2014 года, результаты которых показали, что связанные с рибосомами днРНК отличаются низкой эволюционной консервативностью и содержат гомологи у других видов от 0 до 15,6%, тогда как для БКГ этот показатель более 95% для позвоночных и 70-73% для растений и грибов. Анализ экспрессии днРНК в клетках 6 различных эукариот (A. thaliana, S. cerevisiae, Daniorerio, D. melanogaster, Homosapiens, Musmusculus) видов показал аналогичный кодирующий потенциал и ограничения последовательностей с эволюционно молодыми белками. Полученные данные свидетельствуют о взаимосвязи с рибосомами значительной доли днРНК (от 28,6% у S. cerevisiae до 81,9% у мыши), что свидетельствовало об их трансляции. Было также обнаружено, что эволюционно молодые БКГ, кодирующие экспериментально проверенные белки, характеризовались общими с генами днРНК свойствами. Такие БКГ содержали короткую открытую рамку считывания, занимающую небольшую часть транскрипта, а также имели низкий показатель кодирования, аналогичный таковому для днРНК. Это говорит о происхождении таких эволюционно молодых БКГ от генов днРНК благодаря отбору пептидов, участвующих в адаптивных для выживания организмов процессах [52].
Трансляция некодирующих РНК в функциональные пептиды и белки
Возникновение новых БКГ из генов днРНК у различных животных и растений обусловлено их трансляцией в пептиды, которые принимают участие в разнообразных биологических процессах. Данные о роли днРНК в качестве источников БКГ говорят о возможности образования из транскриптов их генов более крупных молекул полипептидов [49-52]. Это происходит в ходе эволюции образуемых генов с увеличением их размеров благодаря инсерциям МГЭ с их последующей экзонизацией [27, 28]. Помимо эволюционной роли в возникновении БКГ по данным филогенетических исследований, появляется все больше свидетельств непосредственной трансляции днРНК у эукариот. При этом механизмы возникновения более крупных БКГ из всей или большей части последовательности гена днРНК может отличаться от механизмов образования транслируемой молекулы РНК из днРНК, длина которой значительно меньше зрелой днРНК. Так, у мыши и человека обнаружен специфический для скелетных мышц пептид миорегулин (MLN), образуемый из ORF эволюционно консервативной днРНК, которая обозначена для человека как LINC00948, а для мыши как AK009351. У обоих организмов гены днРНК состоят из 3 экзонов и имеют длину более 15000 п.н. В то же время OFR, из которой транслируется пептид MLN, имеет длину лишь 138 п.н. и расположен в 3 экзоне генов. Образуемый пептид регулирует прохождение через мембрану саркоплазматического ретикулума ионов кальция, тем самым управляя расслаблением мышц [46]. Сходным механизмом действия обладает пептид, DWORF, также транслируемый из днРНК и экспрессируемый в тканях миокарда [47]. У растений при трансляции ORF днРНК образуются регулирующие временем цветения пептиды COLDAIR и COOLAIR, важный для симбиоза с бактериями пептид ENOD40 и управляющий поглощением фосфатов белок IPS1 [48].
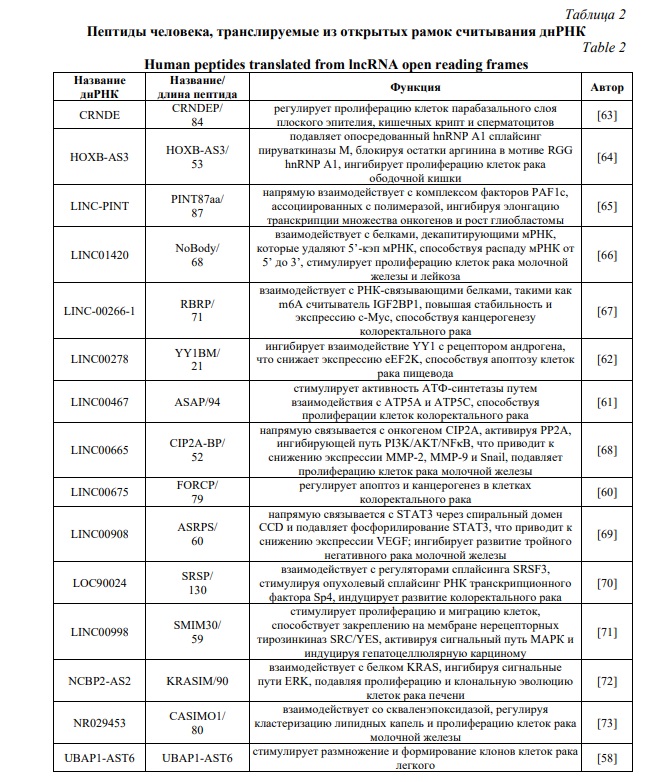
Наибольший интерес представляют исследования трансляции днРНК человека, поскольку образуемые при этом пептиды специфически экспрессируются или подавляются в злокачественных новообразованиях и могут служить объектами для таргетной терапии опухолей (табл. 2). Поскольку МГЭ играют важную роль в возникновении, эволюции и регуляции днРНК, участие образуемых от нкРНК пептидов и белков подтверждает роль МГЭ в канцерогенезе [25]. Можно предположить, что одними из мишеней таких пептидов в опухолевых клетках являются МГЭ, что является предметом дальнейших исследований. Опубликованные в 2019 году результаты полноразмерного секвенирования транслируемых РНК и профилирования рибосом свидетельствуют о том, что 3330 днРНК человека связываются с рибосомами с активной элонгацией трансляции. Протеомный анализ позволил авторам обнаружить 308 новых белков, образуемых в результате трансляции днРНК [58]. Подобные исследования были проведены и другими исследовательскими группами, в которых были определены 128 пептидов, закодированных в последовательностях днРНК [59]. Следует отметить, что некоторые пептиды образуются при трансляции днРНК не в нормальных клетках организма, а только в опухолевых, что свидетельствует механизме эволюции опухолей за счет образования новых БКГ. Это отражает общие эволюционные механизмы, когда отбираются возможные варианты трансляции нкРНК для формирования новых адаптивных свойств живых организмов. Например, днРНК LINC00675 транслируется с образованием малого консервативного белка FORCP из 79 аминокислот, который экспрессируется клетками колоректального рака, тогда как данный протеин не характерен для всех нормальных клеток человека [60]. Для того же типа злокачественного новообразования выявлено образование из днРНК LINC00467 пептида ASAP, который не только экспрессируется в нормальных клетках, но также является консервативным для высших млекопитающих, регулируя функцию митохондрий [61]. Это свидетельствует о развитии различных путей эволюции злокачественных новообразований с использованием как вновь образуемых, так и консервативных пептидов, участвующих в канцерогенезе. Было показано также, что на экспрессию днРНК влияют изменения внутренней среды организма и внешней среды, что свидетельствует о потенциале исследований в данном направлении для выявления факторов риска злокачественных опухолей и возможности их коррекции. Так, на экспрессию пептида YY1BM влияют не только андрогены (в связи с чем он специфично транслируется у мужчин), но также сигаретный дым [62].
Некоторые днРНК являются также прекурсорами для микроРНК, поэтому их кодирующие области в ДНК одновременно являются генами днРНК и генами микроРНК. Более того, продукты транскрипции таких генов также обладают потенциалом транслироваться в функциональные пептиды. Примерами являются днРНК MIR497HG [56], днРНК MIR22HG [54], днРНК MIR155HG [55]. Транскрибируемые из генов микроРНК незрелые при-микроРНК также способны связываться с рибосомами и формировать функциональные пептиды, обозначаемые как miPEPs [74, 75] в связи с наличием в них коротких открытых рамок считывания (smORF), что позволяет им взаимодействовать с рибосомами [74]. Пептиды и белки, кодируемые при-микроРНК, экспрессируются в клетках растений и животных, регулируя рост и развитие нормальных и раковых клеток [76]. Пептиды miPEPs, которые образуются при трансляции при-микроРНК, участвуют в регуляции как БКГ, так и собственных генов микроРНК. Молекулы miPEP характеризуются способностью участвовать в ряде важных биологических реакций у растений, в связи с чем могут быть использованы для улучшения свойств растений. Наилучшим примером является, miPEP172c, который стимулирует трансляцию собственного гена miR172c. В результате этого усиливается формирование узелков в корнях сои с индукцией симбиоза с азотфиксирующими бактериями [74].
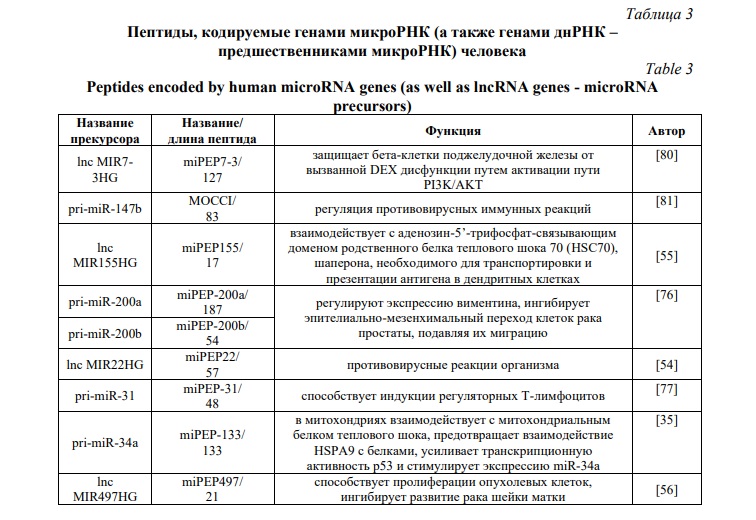
SmORFs, обладающие потенциалом связываться с рибосомами, выявлены у 2% мРНК генов микроРНК растений [75]. Сходными свойствами обладают также процессированные при-микроРНК животных. Был идентифицирован функциональный пептид miPEP31 у человека, образуемый при трансляции pri-miRNA-31, который подавляет экспрессию miR-31, усиливает индукцию регуляторных Т-лимфоцитов, действуя как репрессор транскрипции [77]. Согласно проведенному в 2023 году систематическому обзору научной литературы, в настоящее время известны miPEP различных растений и животных, в том числе miPEP-156a у Brassicarapa, miPEP397a у Brassicaoleacera, miPEP-164b, miPEP165a, miPEP-397a, miPEP858a у Arabidopsis thaliana, miPEP164b у Barbareavulgaris, miPEP-164c, miPEP-171d1, miPEP-172d, miPEP3635b у Vitisvinifera, miPEP171a/b/c/d/e/f у Medicagotruncatula, miPEP171b у Lotusjaponicas, miPEP171i у Oryzasativa, miPEP171e у Solanumlycopersicum, miPEP172c у Glycinemax, miPEP-31 у мыши, miPEP8 у дрозофилы [78]. Недавно описан также miPEP408, участвующий в реакции на мышьяковый стресс и ассимиляцию серы у Arabidopsis [79]. Способностью к трансляции с образованием функциональных пептидов характеризуются не только днРНК и при-микроРНК, но также и другие молекулы известных РНК, такие как малые ядрышковые РНК [59]. Это говорит о грандиозном количестве различных пептидных молекул, которые еще не открыты, функционирующих как специфично для отдельных тканей или органов, так и для патологических процессов, таких как развитие злокачественных новообразований. То есть регуляторные системы геномов эукариот значительно сложнее сложившихся в генетике классических представлений. В таблице 3 содержатся данные об обнаруженных у человека miPEP и их функции.
Функциональность транскриптов белок-кодирующих генов
Интроны БКГ также подвергаются процессингу с образованием функциональных нкРНК [82]. Это подтверждает их эволюционную взаимосвязь с МГЭ, которые являются ключевыми источниками нкРНК [20, 21, 22]. О роли МГЭ в возникновении как днРНК, так и БКГ свидетельствует общее свойство их транскриптов образовывать специфические вторичные и третичные структуры, которые содержат функциональные домены и влияют на собственный процессинг [83], что позволяет предположить их древнее происхождение от МГЭ. Обилие доменов днРНК транспозонными последовательностями связано с тем, что МГЭ образуют пространственные структуры, участвующие в биологических процессах [22]. Биоинформационный анализ позволил установить способность транскриптов БКГ эукариот регулировать собственный сплайсинг, которая зависит от последовательностей нуклеотидов, произошедших в эволюции от МГЭ, в составе их интронов [83]. Данное свойство преемственно от МГЭ, молекулы РНК которых характеризуются формированием шпилек в пространстве, регулирующих распознавание сайтов сплайсинга [84].
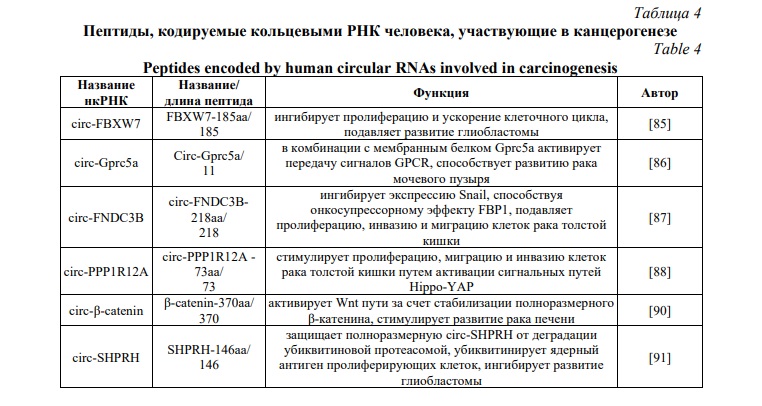
О функциональности молекул мРНК белок-кодирующих генов свидетельствует образование из их интронов специфических кольцевых РНК (кРНК), обладающих множеством регуляторных способностей. Кроме того, кРНК также содержат короткие рамки считывания, связываются с рибосомами и транслируются в пептиды и белки. Кольцевые РНК широко экспрессируются в геномах различных эукариот, поскольку образуются путем обратного сплайсинга, что приводит к образованию ковалентных структур РНК с замкнутой петлей без 3’-5’-полярности. Они не содержат полиадениновых хвостов и экспрессируются на относительно низких уровнях. Подобно днРНК, кРНК могут функционировать в качестве губок микроРНК, участвуют в регуляции развития нервной системы, в механизмах канцерогенеза, экспрессии БКГ [85]. Кроме того, кРНК связываются с промоторами целевых генов, рекрутируя факторы транскрипции, влияя тем самым на транскрипцию БКГ [86]. Наибольший интерес представляют пептиды, кодируемые кРНК у человека в связи с обнаружением их вовлеченности в развитие злокачественных новообразований, поскольку они могут быть использованы в качестве мишеней для противоопухолевой терапии [85-88] (табл. 4). Однако одной из первых кРНК человека, способных к трансляции, стала circ-ZNF609, вовлеченная в регуляцию дифференцировки скелетной мускулатуры. В отличие от белкового продукта основного БКГ (транскрипционный фактор, белок цинковых пальцев), из которого образуется эта кРНК, продукт ее трансляции не содержит доменов цинковых пальцев [89].
Поскольку МГЭ в эволюции являлись источниками возникновения сплайсосомных интронов, а также их изменчивости [24], логично предположить, что способность кРНК транслироваться в функциональные пептиды также отражают универсальные свойства МГЭ и обусловлены возникновением кРНК от транспозонов. Действительно, кРНК, подобно днРНК, характеризуются тканеспецифической экспрессией, а также низкой консервативностью между видами [85-88], что говорит о динамичности их эволюции с помощью перемещений МГЭ. Было показано, что среди всех комплементарных последовательностей, SINE (особенно Alu) человека вносят наибольший вклад в образование кРНК и их разнообразие. Соответственно, разное распределение МГЭ между видами приводит к увеличению сложности экспрессии кРНК при эволюции видов [92]. Это обусловлено ролью Alu в качестве источников акцепторов сплайсинга и ингибиторов трансляции мРНК. Alu являются основными мишенями фермента ADAR, редактирующего РНК, а образование экзонов Alu подавляется рибонуклеопротеином HNRNPC. Было обнаружено, что ядерная РНК-хеликаза DHX9 специфически связывается с инвертированными повторами Alu в составе мРНК белок-кодирующих генов. Увеличение количества содержащих Alu генов, образующих кРНК, происходит при потере DHX9, который действует в качестве резольвазы ядерной РНК, нейтрализующей угрозу нарушений транскрипции и трансляции вследствие вставок Alu [93]. Было предложено, что кРНК длиной 100-1000 нуклеотидов, образующиеся при участии малых саморасщепляющихся РНК, которые обозначаются как рибозимы типа «молот», закодированы неавтономными РЭ ретрозимами [94]. Это новое семейство РЭ оказалось широко распространенным в геномах эукариот. Образуемые в результате экспрессии этих РЭ транскрипты, содержащие мотивы типа «молот», легко обнаружить с помощью РНК-блоттинга [95]. Не исключено, что широкое распространение Alu в геномах эукариот (занимают 11% генома человека) обусловлено свойством образовывать кРНК, которые участвуют в регуляции экспрессии генов в ходе адаптивной эволюции организмов. Действительно, для формирования кРНК необходимо спаривание нуклеотидов РНК между последовательностями, фланкирующими сайты обратного сплайсинга, благодаря наличию Alu в составе интронов генов [96].
Заключение. В данной статье представлены данные, подтверждающие роль МГЭ в качестве универсальных единиц эволюции, свойства которых к противоборству с образованием систем процессинга стало основой многофункциональности их продуктов транскрипции и трансляции. Описана роль МГЭ в возникновении новых БКГ, генов микроРНК, днРНК и кРНК в эволюции. Представлены сведения об образовании 50 БКГ только от транспозазы ДНК-транспозонов в эволюции у различных эукариот, в том числе 16 генов у человека, а также возникновение различных БКГ от генов ретроэлементов. Одним из преимуществ одомашнивания генов МГЭ геномами эукариот является вовлеченность образуемых мРНК в регуляторные адаптивные сети, а также функциональность самих молекул мРНК после их процессинга. Способность транскриптов генов днРНК и микроРНК к трансляции отражает свойство МГЭ образовывать одновременно функциональные РНК и их белковые продукты. Исследование данных свойств наиболее перспективно в современной онкологии, поскольку появляется все больше данных о вовлеченности, образуемых из днРНК, при-микроРНК и кРНК пептидов в канцерогенезе. Сами молекулы этих нкРНК могут быть использованы в качестве мишеней для воздействия, а их пептидные продукты трансляции как инструменты. В статье представлены данные об образовании 15 участвующих в канцерогенезе специфических функциональных пептидов в результате трансляции днРНК, 4 пептидов – из при-микроРНК и 6 пептидов – из кРНК.
Информация о финансировании
Финансирование данной работы не проводилось





















Список литературы